文章标题
2017-11-27 21:30
225 查看
DataSciBowl2017_9th【译文】
原文链接:https://eliasvansteenkiste.github.io/machine%20learning/lung-cancer-pred/作者团队:Deep Breath
项目开源地址:https://github.com/EliasVansteenkiste/dsb3
原文作者:Elias Vansteenkiste @SaileNav
作者单位和职位:比利时根特大学 计算机系统实验室 博士后研究员
作者LinkedIn:https://www.linkedin.com/in/elias-vansteenkiste-33060839/
团队成员:Andreas Verleysen @resivium ;Elias Vansteenkiste @SaileNav ;Fréderic Godin @frederic_godin ;Ira Korshunova @iskorna ;Jonas Degrave @317070 ;Lionel Pigou @lpigou ;Matthias Freiberger @mfreib
0. 海底捞针
要确定一个人是否会在不久的将来罹患肺癌,我们必须确定这个人体内是否存在早期形态的恶性肿瘤。而在患者肺部CT扫描中发现恶性结节就如同大海捞针一般困难。为了支撑这个论点,我们随机取了LIDC/IDRI数据集中某个病例,这些数据是从Luna16中获得的。我们选择使用了这个数据集(以下简称Luna16),因为这个数据集包含放射科医师的详细注释。

[align=center]LUNA数据集中某个恶性结节的三个方向特写(X方向-左、Y方向-中间 和 Z方向-右)[/align]
Luna16数据集中恶性结节的平均半径为4.8mm,典型的CT扫描采集的体积为400mm×400mm×400mm。半径比大约为1:1000000,所以我们的任务是设计检测一个比输入量小一百万倍目标的算法。而且这也决定了我们整个任务的输入是一个这么大体积的CT。分析CT扫描对于放射科医师来说是一个巨大的负担,对于只使用常规卷积神经网络的分类算法来说同样是一个困难的任务。更坑爹的地方在于我们必须从Kaggle官方提供的数据集中预测一年内某个患者罹患癌症的概率。Luna16数据集中包含已被诊断罹患肺癌的患者,但是在Kaggle提供的数据集中,一年内被诊断患癌的病人其CT扫描中致癌的结节可能尚未发展成恶性结节。因此,直接将Kaggle提供的数据和标签进行训练是不合理的,但我们也尝试过直接训练,由于观察到网络的表现不尽人意,所以有了以下几个步骤。
1. 结节检测
1.1 候选结节分割
为了减少输入到分类网络中的信息量,首先尝试分割出候选肺结节。我们设计了一个网络来分割CT切片中的结节,Luna16数据集中包含每个结节的注释(位置、损伤最大程度的截面中的病灶最长直径),我们使用这些数据来训练我们的分割网络。CT扫描来自不同的扫描仪,其层厚层距等都有一定的差异,我们将所有的CT扫描重新调整尺寸,使得每个体素都代表1x1x1立方毫米的小立方体。对于每个结节立方体,其GroundTruth是一个32x32x32mm的二进制掩模,其每个体素表示体素是否在结节内。掩模通过使用annotations.csv文件中的直径和坐标来构造。我们选择改进后的Dice系数作为损失函数。Dice系数是图像分割领域常用的度量标准,其用于解决早期癌症检测很重要的小结节训练时正负样本不均衡问题非常有帮助。上文提到病人结节所占的体素个数与其补集相比约为1:1000000,这是一个典型的正负样本不均衡的问题。使用Dice系数的缺点是如果GroundTruth中没有结节,则它值恒为0,所以喂入网络中的立方体至少存在一个结节。我们以结节质心为中心切取出一个64x64x64的立方体,为了增加样本的多样性我们使用了全旋转和轻微偏移等数据增广操作。设定平移和旋转参数时尽量使结节留在结节质心附近的32x32x32立方体内。
U-net架构是2D图像分割的通用架构,我们设计的网络主要基于这个架构。我们使用它的思想并将输入变为3Dtensor,网络主要由具有无填充的3x3x3滤波器内核的卷积层构成。架构中只有一个Maxpool,我们尝试了更多的Maxpool,但是实验结果证明效果没有什么提升,也许是由于我们的分割网络输入维度为64x64x64,对于U-net的输入张量维度572x572来说分辨率太低。
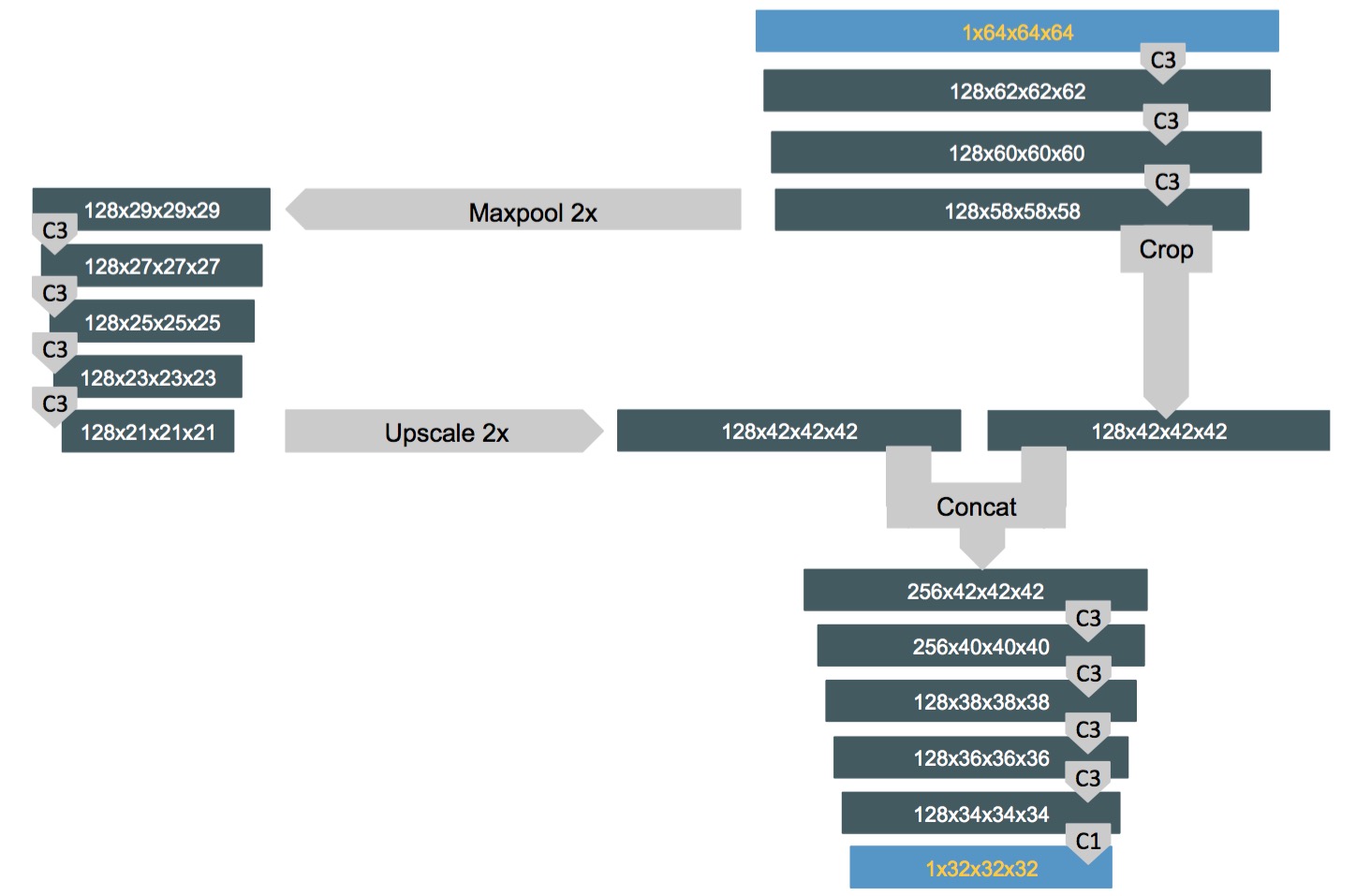
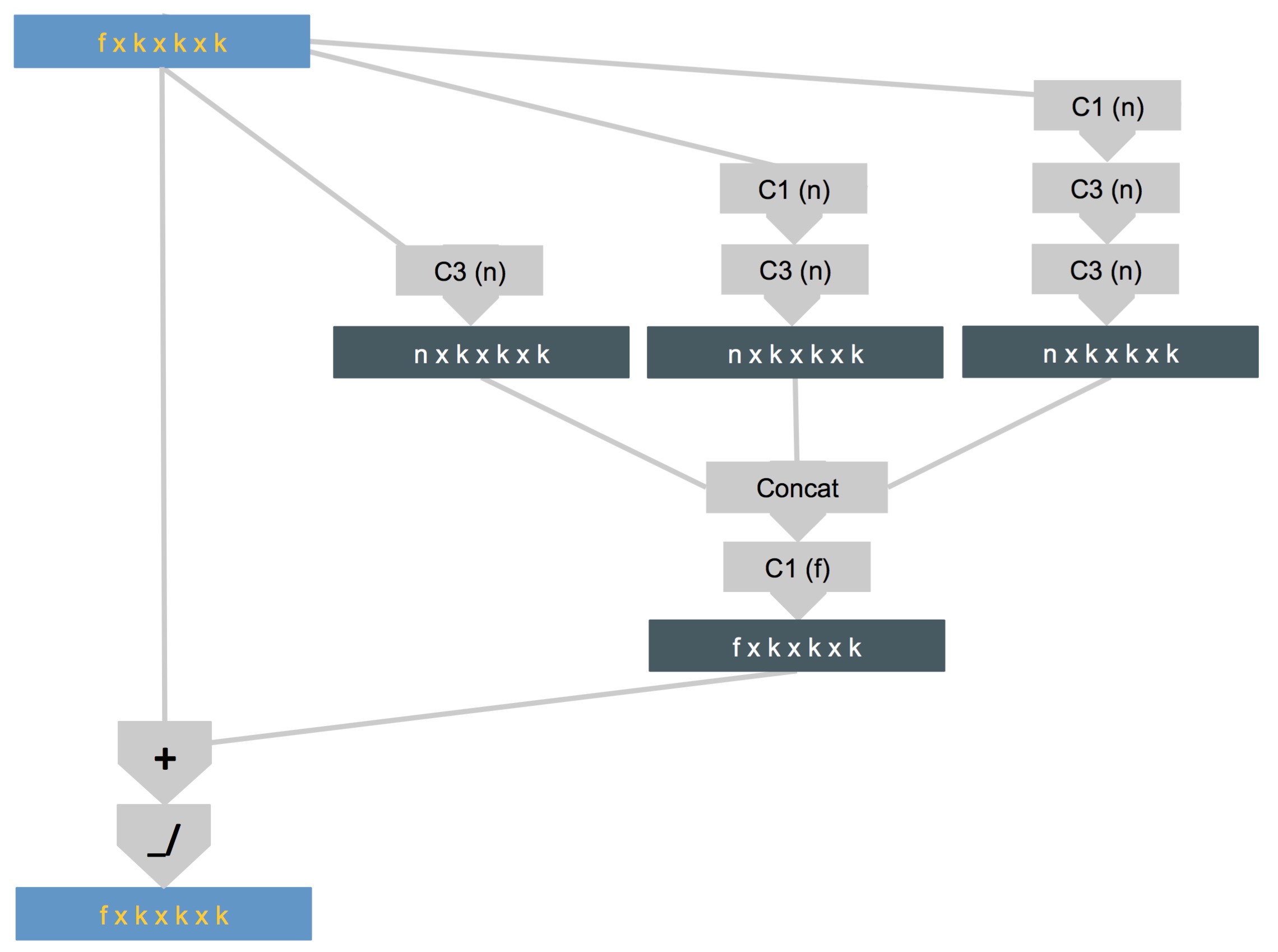
[align=center]分割网络架构的原理图[/align]
注:Tensor维度在深灰色框内显示,网络操作在浅灰色框内。C1是具有1x1x1滤波器内核的卷积层,C3是具有3×3×3滤波器内核的卷积层。
训练好的网络用于分割Luna16和DataSciBowl2017数据集中患者的整张CT,从CT扫描中以32x32x32为步长切出大小为64x64x64的立方体并将其喂入结节分割网络,网络的输出是每个体素处于结节内部的概率。
1.2 候选结节检测
上一步我们已经对肺部CT中的每个体素进行了一个概率预测,现在我们需要找出每个候选结节的中心。通常可通过寻找概率高的体素斑点可以发现结节中心,一旦发现了这样的高概率斑块,他们的中心将被用作候选结节的中心。我们使用了高斯差分(DOG)的方法来检测斑块,因为DOG使用了类似于拉普拉斯算子计算量少的方法。我们调用了skimage包中的函数。在检测斑块之后,我们生成了一个表格,这个列表上有大量的候选结节,DataSciBowl2017提供的数据经斑块检测后平均每个病人的候选结节个数为153。这说明有大量的假阳性结节存在,所以我们针对这个问题做了如下两个工作:斑块检测之前筛除肺实质之外的结节;
训练一个结节分类网络来进一步筛除假阳性结节。
2. 假阳性减少
2.1 肺实质分割
由于上述肺结节分割网络无法根据上下文来作预测,因此在肺实质外部也产生了许多假阳性结果。为了解决这个问题,我们决定自己设计一个肺实质分割的算法以筛除肺实质外的假阳性结节。最开始我们使用类似于Kaggle教程中的方法,它是使用一些简单阈值处理和形态学操作来分割肺实质。我们粗略浏览了分割效果之后注意到这个方法的效率对肺实质大小依赖过大,即常规切片上会工作良好,每当有两个以上的小空腔时,效果就大打折扣。所以我们最终采用了3D分割的方法,主要思路是使用3D的方法从围绕肺实质的凸包中分割出非肺实质区域。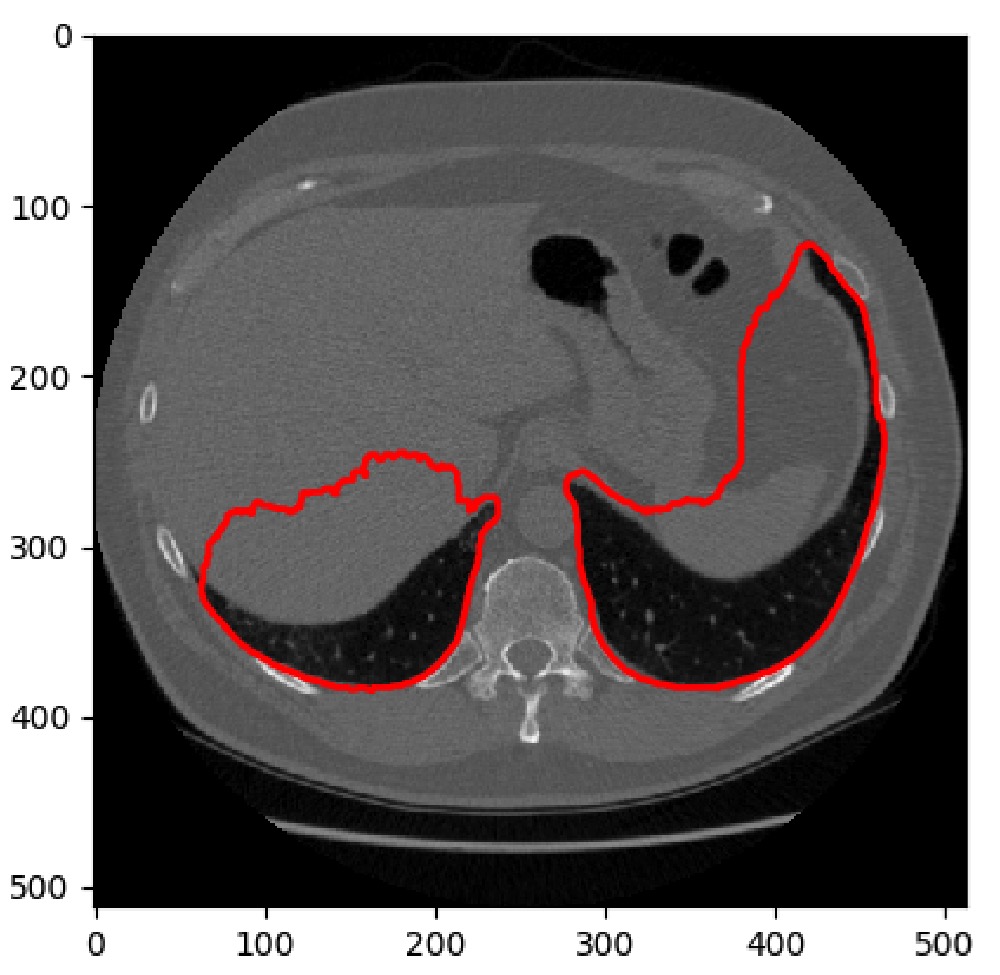
[align=center]两个空腔以上效果图,只分割出了两个大的[/align]

[align=center]使用形态学方法(左)和我们的方法(右)的效果对比图[/align]
2.2 肺结节分类
2.2.1 数据准备
为了进一步筛除肺实质内部候选结节中的假阳性结节,我们尝试训练一个结节分类网络来预测斑块检测后的候选结节是否为阳性结节。Luna16提供了一个csv文件,为每个病人给出了一个假阳性和阳性结节的列表,所以我们使用阳性和假阳性标注文件来训练一个结节分类网络。最终针对每个结节都提取了48x48x48立方体来训练结节分类网络,并在±3mm范围内做全旋转和轻微偏移的数据增强操作以获得更多样本。2.2.2 网络设计
如果我们想要设计的网络既能检测到直径小于3mm的小结节,同时也能检测到直径大于30mm的结节,网络架构应该能以非常窄和宽的感受野来提取特征。Inception-resnet v2(膜拜Kaiming大大)架构非常适合在不同的感受野上训练特征。我们的网络很大程度上参考了这个网络结构,通过简化inception-resnet v2并将其思想迁移到了3Dtensor上,我们提取了可复用的模块,并测试了多种不同网络结构的效果。* 空间压缩模块,对输入tensor的维度通过不同的方法进行压缩,如图所示,分别是最大值池化与一系列不同的卷积操作。简单介绍下图中的图例,C3(f/4,S2)就是卷积核为3×3×3,步长为2,输出通道数为f/4,没标步长的步长均为1。
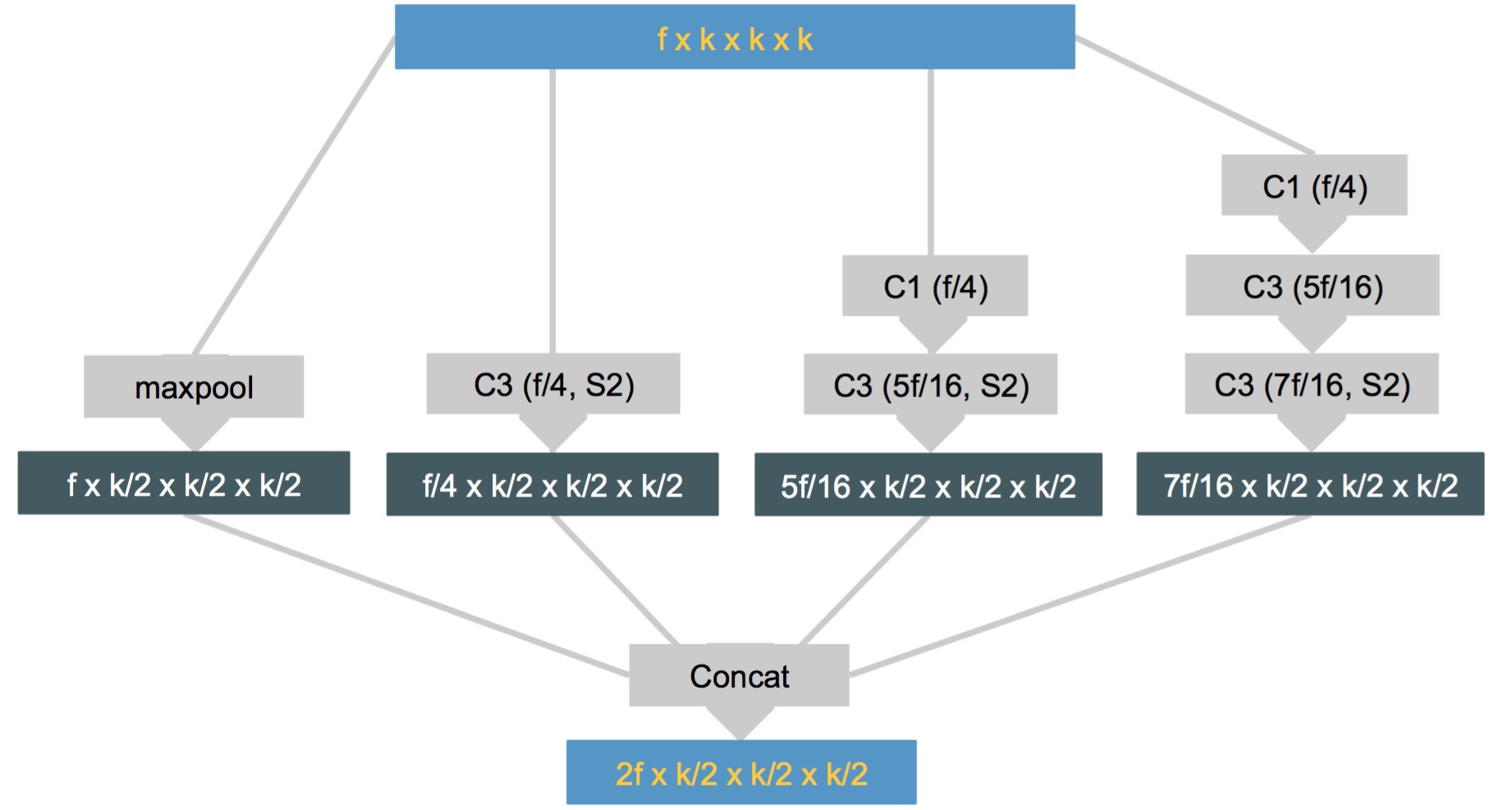
[align=center]空间压缩模块(spatial_red_block)[/align]
* 特征压缩模块,这个模块比较简单,就是用卷积核为1×1×1的卷积操作压缩特征的维度,卷积核的数量(即输出通道数)是输入通道数的一半。
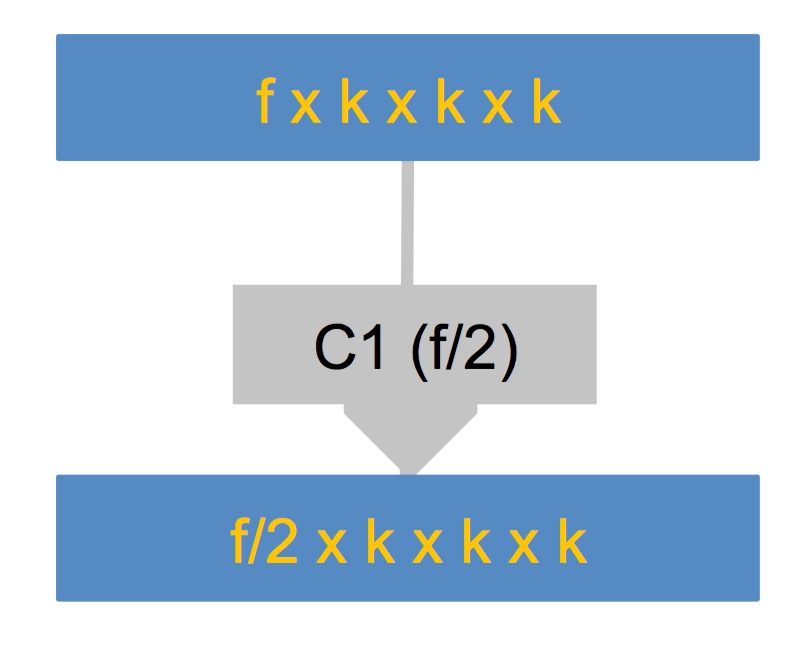
[align=center]特征压缩模块(feat_red)[/align]
* 残差模块,为了便于理解,大家请先阅读下感受野及其计算,为了使模型可以在不同的感受野上训练特征,我们采用三个不同的卷积操作(卷积核大小不同,卷积层数量不同),最浅的卷积操作只有一层,卷积核为1×1×1,因此该卷积操作并没有增大感受野。最深的卷积操作有三层,通过三次卷积,将感受野扩到到5×5×5。三种不同的卷积操作输出的特征图concat在一起(在channel维度上concat),并通过一个卷积核为1×1×1的操作将特征维度(其实还是通道数)与输入特征维度相匹配。将该模块的输出与模块的输入直接相加(resnet经典结构大家应该都不陌生吧),然后通过Relu进行非线性映射。
[align=center]残差模块(res_conv_block)[/align]
介绍完三个主要的模块,接下来的任务是确定模块的数量与结构,通过实验,我们发现如下所示的结构表现得比较好:
def build_model(l_in): l = conv3d(l_in, 64) l = spatial_red_block(l) l = res_conv_block(l) l = spatial_red_block(l) l = res_conv_block(l) l = spatial_red_block(l) l = res_conv_block(l) l = feat_red(l) l = res_conv_block(l) l = feat_red(l) l = dense(drop(l), 128) l_out = DenseLayer(l, num_units=1, nonlinearity=sigmoid)//sigmoid激活函数将输出映射到0-1之间(感觉输出2通道softmax一下更常用啊orz) return l_out
Note:与Inception-resnet v2模型相比,我们的模型在输入前端只有一层卷积层(代码第二行),而Inception-resnet v2有若干层(5个卷积层与2个maxpooling层)用来减少输入图像空间维度。
2.2.3 结果
我们的验证集由118个总共238个结节的患者组成,数据来源于Luna16。在结节分割和斑点检测后,检测出了238个结节中的229个,但是我们有大约17000个假阳性结节。为了减少假阳性,候选结节按照结节分类网络给出的预测值进行排名后,以下是我们取不同阈值所得的数据。| 取Top n% | 阳性结节数 | 假阳性结节数 |
|---|---|---|
| 10 | 211 | 959 |
| 4 | 187 | 285 |
| 2 | 147 | 89 |
| 1 | 99 | 19 |
3. 结节良恶性预测
其实我觉得做个良恶性判断之后再提交成绩可能会更好一点,因为可以进一步筛除假阳性结节,不过如果不让使用外部数据的话,这步好像没法做。4. 患者肺癌预测
这一步是DataSciBowl2017比赛中的要求,和天池医疗AI大赛相关关系不是特别大,有兴趣的同学可以去读读原文。4.1 迁移学习
4.2 根据结节预测
4.3 联合预测
疑虑与思考
分割网络输入输出—输入:每个阳性结节中心外部的64x64x64立方体;输出是32x32x32,那对应的GT是直接切一块32x32x32的还是切出64x64x64之后resize成32x32x32的?(已发送邮件咨询原作,有看懂的大神还望不吝赐教)之前打算着整理好原作的脚本的,这些天手里的机器在跑别的东西,再加上原作代码有点乱,就一直拖着。大家有整理好的欢迎开帖子,这里无偿帮你添加传送门。
译者1:lilhope(结节分类部分)
译者2:JChen(其他部分)
如果有什么疑问,欢迎在下方留言,这样大家都看得到关于这个问题的讨论。
