RNA-seq数据处理流程(以胶质瘤数据为例)
2017-08-31 14:15
465 查看
一、下载比对参考基因组文件,为HISAT2配置index
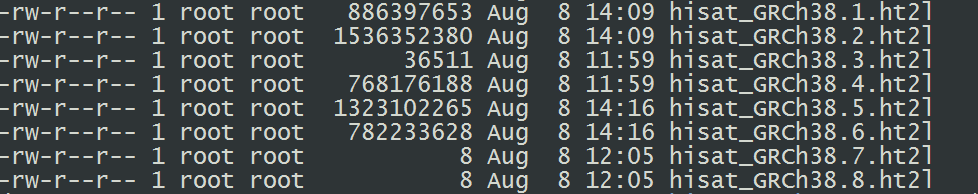
配置index需要基因组注释文件(通常为gtf格式)以及基因组序列文件(fasta格式)。多个数据库提供此注释文件,此处采用ensemble提供的文件。# 从ensemble中下载最新版本的人类基因组注释文件(gtf格式) wget ftp://ftp.ensembl.org/pub/release-89/gtf/homo_sapiens/Homo_sapiens.GRCh38.89.gtf.gz # 下载人类基因组序列 wget ftp://ftp.ensembl.org/pub/current_fasta/homo_sapiens/dna_index/Homo_sapiens.GRCh38.dna.toplevel.fa.gz #配置HISAT2的index hisat2-build -p 8 Homo_sapiens.GRCh38.dna.toplevel.fa GRCh38_ensembl_dna 1>build_index.log& #配置index用时约2小时,结果文件为下图所示
二、下载sra数据
进入GEO页面输入id号,进入sra study的ftp下载页面,复制sra文件的链接,在linux下执行以下命令进行下载。
nohup wget -c [文件链接] > download.log&
三、将sra文件转换成fastq.gz格式
每秒可生产1M文件,工具不支持多线程。#对文件夹下的所有sra文件批量处理 for i in *sra do echo $i # 对于双端测序数据,需要加--split-3参数,每样本处理约10min fastq-dump --split-3 $i --gzip done
每个sra文件会产生两个
fastq.gz文件,名称分别为
*_1.fastq.gz和
*_2.fastq.gz
四、对fastq数据进行质控
使用fastqc(http://www.bioinformatics.babraham.ac.uk/projects/fastqc/)进行质量控制,代码如下:for id in *fastq do echo $id # 此处使用8线程,平均每文件处理约10min /home/RNAseq_tool/FastQC/fastqc -t 8 $id -o /data/GSE86202/0.fastqc/ done
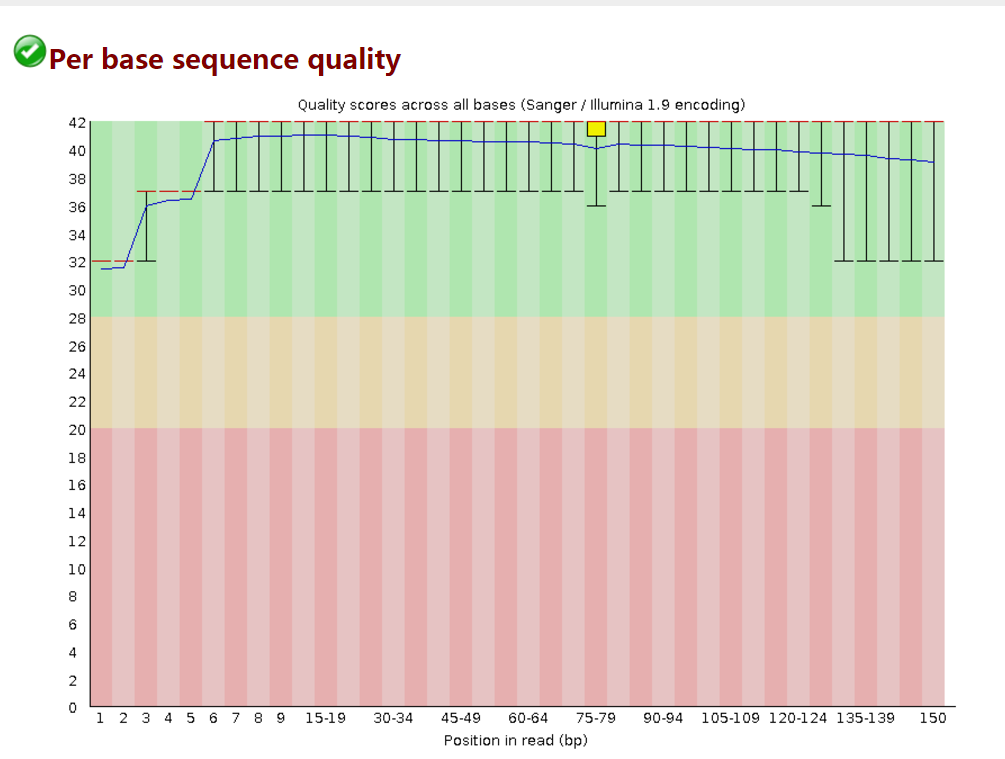
得到一个html格式报告以及包含html及表格形式报告的压缩包。其中html文件可以看出数据质量。以
SRR4095543_1.fastq为例,下图是其原始序列质量,可看出测序质量较高。
但是其在5’端存在adapter,从下图可以看出。
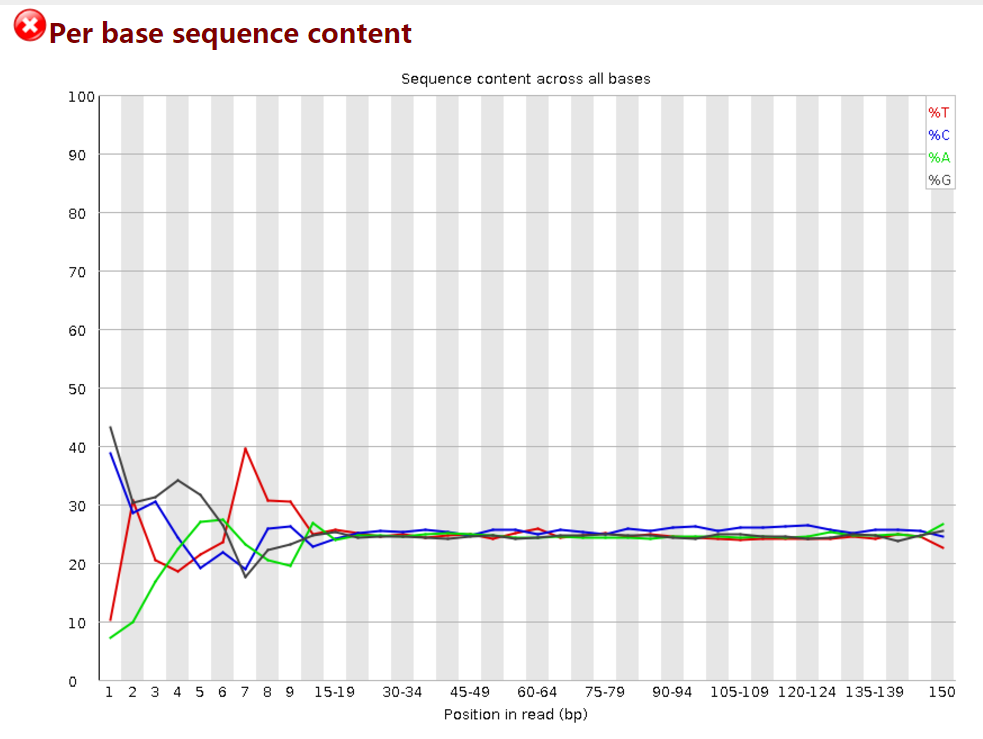
因此需要切除5’端接头,此处选择切除10bp。
五、接头处理并再次质控
使用cutadapt(https://pypi.python.org/pypi/cutadapt/1.2.1)进行接头处理,代码如下,代码位于服务器/data/GSE86202/1.cutadapt/cutadapt.sh
for id in *fastq do echo $id # 切除序列5'前10个碱基,每个文件处理约5min cutadapt -u 10 -o /data/GSE86202/cut_$id $id done
注意:在实际流程中,原始数据可能存在各种各样的问题,需要根据fastqc质控结果按需处理。本例中的处理方式仅对本数据有效。
再次质控结果:
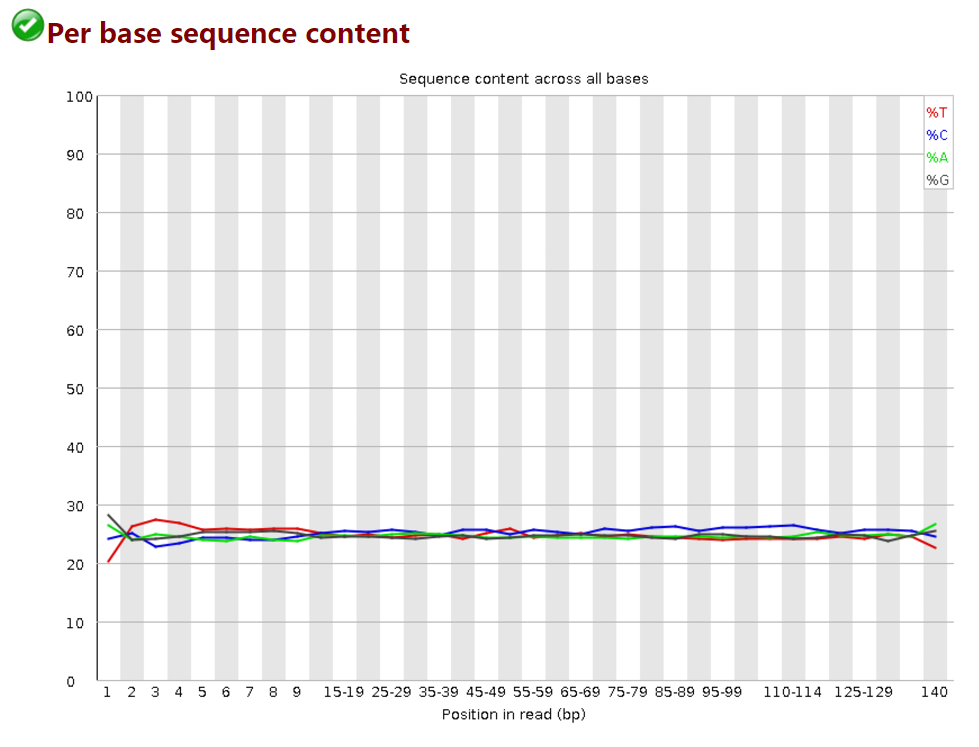
可以看到每个碱基的碱基组成趋于正常。
六、序列比对
本例使用HISAT2进行序列比对,其速度更快且精度更高,是Tophat的优秀替代工具。比对代码如下。DATA_PATH=/data/GSE86202/1.cutadapt
REF_PATH=/data/reference_data
OUT_PATH=/data/GSE86202/4.hisat2
FILE=/data/GSE86202/filelist.txt
cd $DATA_PATH
cat $FILE | while read line
do
hisat2 -x $REF_PATH/hisat_GRCh38 --no-mixed -1 $DATA_PATH/cut_${line}_1.fastq -2
#将HISAT2处理的结果输出到samtools转化为bam格式
#此处使用6核,约使用6.4G内存,平均每文件处理需30min
$DATA_PATH/cut_${line}_2.fastq -p 6 |samtools view -bS 1>$OUT_PATH/${line}.bam
done七、对bam文件排序
使用samtools(http://samtools.sourceforge.net/)对bam文件进行排序并添加indexFILE_PATH=/data/GSE86202/4.hisat2
OUT_PATH=/data/GSE86202/5.samtools
cd $FILE_PATH
for file in *.bam
do
# 对bam文件进行排序(-n参数必须,表示按照read name进行排序)
samtools sort -n $FILE_PATH/${file} -o $OUT_PATH/sorted_${file} -@ 6
# 对已经排序的bam文件进行简单质量控制
samtools flagstat $OUT_PATH/sorted_${file} -@ 6 > $OUT_PATH/${file}.stat
done
# 质控后得到结果如下图所示
可看到比对结果良好。
八、使用htseq得到count值
使用RSeQC进行对bam文件的简单质控和各项参数的检查FILE_PATH=/data/GSE86202/5.samtools/gencode_genome
REF_PATH=/data/reference_data/gtf
OUT_PATH=/data/GSE86202/10.htseq/gencode_genome
cd $FILE_PATH
for i in `seq 42..47`
do
nohup htseq-count -t exon -s reverse \
-r name -f bam $FILE_PATH/name_new_SRR40955${i}.bam \
$REF_PATH/gencode.v26.annotation.gtf \
> $OUT_PATH/name_new_SRR40955${i}.bam.count \
> SRR40955${i}_count.log 2>&1 &
done得到count值文件数与样本数据数量相等,为两列值(如下图所示),其中包括了测序数据覆盖gene的ensemble号及count值。
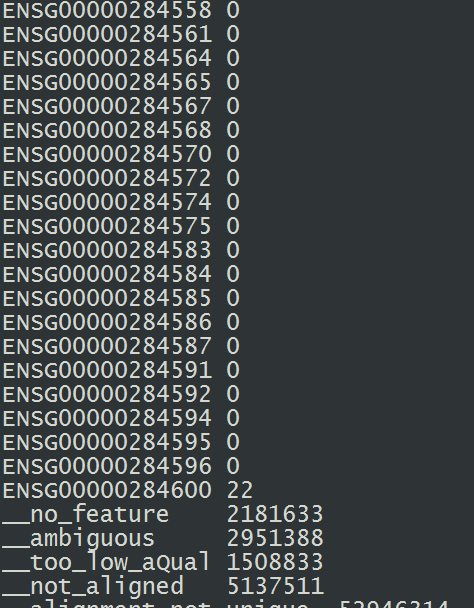
写perl脚本
combine_count.pl将所有count值进行合并,
合并文件如下图所示:

九、使用R及count矩阵进行差异分析
count矩阵数据可以直接使用R中DEseq2包进行差异分析、GO分析以及pathway分析。
相关文章推荐
- 用于自动处理高通量测序(RNA-seq)数据的R脚本,2015年5月6日更新
- 数据处理流程
- RNA-Seq数据分析
- 磁共振实验数据SPM8处理流程(继续)
- CMDB处理数据流程
- 客户余家琪反馈数据有误处理流程记录
- PHP手机归属地查询流程及数据获取格式化处理
- 基于命令行的mahout软件0.8版本Canopy算法分析的数据处理流程
- ROI_PAC自带测试数据处理流程
- RNA-Seq数据去接头(Adapter)
- 【EMV L2】静态数据认证处理流程
- 数据处理流程
- 阿里云数据处理流程
- TBC处理静态数据流程
- 在JSP 添加表单数据到本地数据库的处理流程小结
- CPU处理数据流程
- 基因数据处理19之BWA匹配算法串产生、匹配、评价等整体流程
- libnids-1.21 中 IP 分片重组分析 之数据结构与处理流程
- Hadoop和Hive的数据处理流程
- RNA-seq流程报告
