遗传算法入门例子和总结
2017-05-05 20:47
471 查看
遗传算法的手工模拟计算示例
为更好地理解遗传算法的运算过程,下面用手工计算来简单地模拟遗传算法的各
个主要执行步骤。
例:求下述二元函数的最大值:
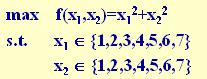
(1) 个体编码
遗传算法的运算对象是表示个体的符号串,所以必须把变量 x1, x2 编码为一种
符号串。本题中,用无符号二进制整数来表示。
因 x1, x2 为 0 ~ 7之间的整数,所以分别用3位无符号二进制整数来表示,将它
们连接在一起所组成的6位无符号二进制数就形成了个体的基因型,表示一个可
行解。
例如,基因型 X=101110 所对应的表现型是:x=[ 5,6 ]。
个体的表现型x和基因型X之间可通过编码和解码程序相互转换。
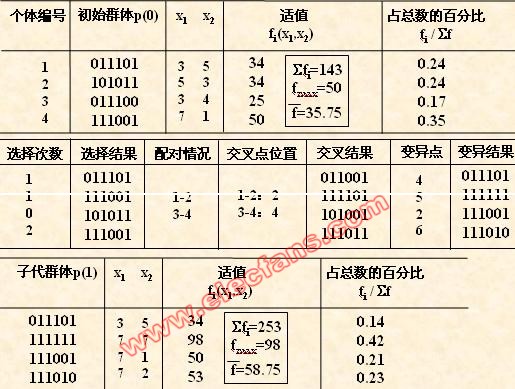
(2) 初始群体的产生
遗传算法是对群体进行的进化操作,需要给其淮备一些表示起始搜索点的初始
群体数据。
本例中,群体规模的大小取为4,即群体由4个个体组成,每个个体可通过随机
方法产生。
如:011101,101011,011100,111001
(3) 适应度汁算
遗传算法中以个体适应度的大小来评定各个个体的优劣程度,从而决定其遗传
机会的大小。
本例中,目标函数总取非负值,并且是以求函数最大值为优化目标,故可直接
利用目标函数值作为个体的适应度。
(4) 选择运算
选择运算(或称为复制运算)把当前群体中适应度较高的个体按某种规则或模型遗传到下一代群体中。一般要求适应度较高的个体将有更多的机会遗传到下一代
群体中。
本例中,我们采用与适应度成正比的概率来确定各个个体复制到下一代群体中
的数量。其具体操作过程是:
• 先计算出群体中所有个体的适应度的总和 fi ( i=1.2,…,M );
• 其次计算出每个个体的相对适应度的大小 fi / fi ,它即为每个个体被遗传
到下一代群体中的概率,
• 每个概率值组成一个区域,全部概率值之和为1;
• 最后再产生一个0到1之间的随机数,依据该随机数出现在上述哪一个概率区
域内来确定各个个体被选中的次数。
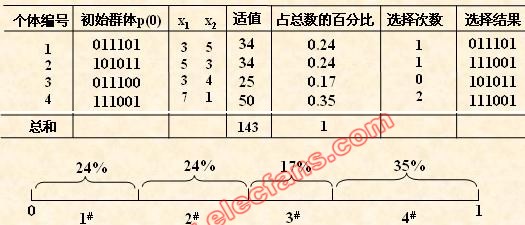
(5) 交叉运算
交叉运算是遗传算法中产生新个体的主要操作过程,它以某一概率相互交换某
两个个体之间的部分染色体。
本例采用单点交叉的方法,其具体操作过程是:
• 先对群体进行随机配对;
• 其次随机设置交叉点位置;
• 最后再相互交换配对染色体之间的部分基因。
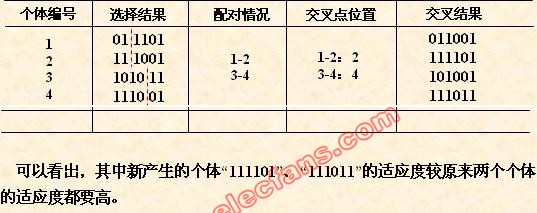
(6) 变异运算
变异运算是对个体的某一个或某一些基因座上的基因值按某一较小的概率进
行改变,它也是产生新个体的一种操作方法。
本例中,我们采用基本位变异的方法来进行变异运算,其具体操作过程是:
• 首先确定出各个个体的基因变异位置,下表所示为随机产生的变异点位置,
其中的数字表示变异点设置在该基因座处;
• 然后依照某一概率将变异点的原有基因值取反。
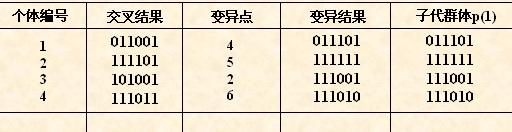
对群体P(t)进行一轮选择、交叉、变异运算之后可得到新一代的群体p(t+1)。
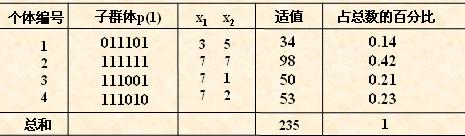
从上表中可以看出,群体经过一代进化之后,其适应度的最大值、平均值都得
到了明显的改进。事实上,这里已经找到了最佳个体“111111”。
[注意]
需要说明的是,表中有些栏的数据是随机产生的。这里为了更好地说明问题,
我们特意选择了一些较好的数值以便能够得到较好的结果,而在实际运算过程中
有可能需要一定的循环次数才能达到这个最优结果。
四.C语言实现
#include <stdio.h>
#include <stdlib.h>
#include <time.h>
/////////////The definiton of user data////
#define Cmax 100 //certain maximal value
#define Cmin 0 //certain minimum value
#define LENGHT1 3 //the chromosome length of 1st variable
#define LENGHT2 3 //the chromosome length of 2nd variable
//总染体长度
#define CHROMLENGTH LENGHT1+LENGHT2
const int MaxGeneration = 100; //最大代数
const int PopSize = 10; //样本大小
const double Pc = 0.6; //交叉概率
const double Pm = 0.001; //变异概率
////////////// 数据结构定义///////////////////
struct Individual{
char chrom[CHROMLENGTH + 1]; //一个个体的染色体
double value; //目标值
double fitness; //适应度
};
int generation ; //进化次数
int bestIndex; //最好个体的下标
int worstIndex; //最坏个体的游标
Individual bestIndividual ; //当前一代中的最好个体
Individual worstIndividual ; ///当前一代中的坏个体
// best individual by now
Individual currentBest ;// 到目前为止的最好个体
Individual population [PopSize] ;//样本
///////////////////////
void generateInitialPopulation();
void generateNextPopulation();
void evalutePopulation();
long decomdeChromosome(char*, int, int);
void calculateObjectValue();
void calculateFitnessValue();
void findBestAndWorstIndividual();
void performEvolution();
void selectionOperator();
void
109bf
crossoverOperator();
void mutationOperator();
void outputTextReport();
//////////////////////////////////////////////
int main(){
generation = 0;
generateInitialPopulation();
evalutePopulation();
while (generation < MaxGeneration) {
generation++;
generateNextPopulation();
evalutePopulation();
performEvolution();
outputTextReport();
}
return 0;
}
//////////////////////////////////////////////////////////////
//////产生第一代样本/////
void generateInitialPopulation() {
int i, j;
srand((unsigned)time(NULL));
for (i = 0; i < PopSize; i++) {
for (j = 0; j < CHROMLENGTH; j++) {
population[i].chrom[j] = ((rand() % 10) < 5) ? '0' : '1';
}
population[i].chrom[CHROMLENGTH] = '/0';
}
}
////////产生下一代样本 //////
void generateNextPopulation() {
selectionOperator();
crossoverOperator();
mutationOperator();
}
//变异算子//
void mutationOperator() {
int i, j;
double p;
// bit mutation
for (i = 0; i < PopSize; i++) {
for (j = 0; j < CHROMLENGTH; j++) {
p = rand() % 1000 / 1000.0;
if (p < Pm) {
population[i].chrom[j] = (population[i].chrom[j] == '0') ? '1': '0';
}
}
}
}
//交叉算子///
void crossoverOperator() {
int i, j;
int index[PopSize];
int point, temp;
double p;
char ch;
for (i = 0; i < PopSize; i++) {
index[i] = i;
}
for (i = 0; i < PopSize; i++) {
point = rand() %(PopSize - i);
temp = index[i];
index[i] = index[point + i];
index[point + i] = temp;
}
for (i = 0; i < PopSize - 1; i+=2) {
p = rand() % 1000 / 1000.0;
if (p < Pc) {
point = rand()% (CHROMLENGTH - 1) + 1;
for (j = point; j < CHROMLENGTH; j++) {
ch = population[index[i]].chrom[j];
population[index[i]].chrom[j] = population[index[i + 1]].chrom[j];
population[index[i + 1]].chrom[j] = ch;
}
}
}
}
///选择算子/////////////
void selectionOperator() {
int i, index;
double p, sum = 0.0;
double cfitness[PopSize];
Individual newpopulation[PopSize];
for (i = 0; i < PopSize; i++) {
sum += population[i].fitness;
}
for (i = 0; i < PopSize; i++) {
cfitness[i] = population[i].fitness / sum;
}
// calculate cumulative fitness
for (i = 1; i < PopSize; i++) {
cfitness[i] = cfitness[i] + cfitness[i - 1];
}
for (i = 0; i < PopSize; i++) {
p = rand() % 1000 / 1000.0;
index = 0;
while (p > cfitness[index]) {
index++;
}
newpopulation[i] = population[index];
}
for (i = 0; i < PopSize; i++) {
population[i] = newpopulation[i];
}
}
/////依据某些公式对样本进行评价////
void evalutePopulation() {
calculateObjectValue();
calculateFitnessValue();
findBestAndWorstIndividual();
}
//找出到目前为止最好的个体//////
void findBestAndWorstIndividual() {
int i;
double sum = 0.0;
bestIndividual = population[0];
worstIndividual = population[0];
for (i = 0; i < PopSize; i++) {
if (population[i].fitness > bestIndividual.fitness) {
bestIndividual = population[i];
bestIndex = i;
} else if (population[i].fitness < worstIndividual.fitness) {
worstIndividual = population[i];
worstIndex = i;
}
sum += population[i].fitness;
}
if (generation == 0) {
currentBest = bestIndividual;
} else {
if (bestIndividual.fitness > currentBest.fitness) {
currentBest = bestIndividual;
}
}
}
//计算适应度///
void calculateFitnessValue() {
int i;
long temp1, temp2;
double x1, x2;
for (i = 0; i < PopSize; i++) {
temp1 = decomdeChromosome(population[i].chrom, 0, LENGHT1);
temp2 = decomdeChromosome(population[i].chrom, LENGHT1, LENGHT2);
x1 = temp1 * temp1;
x2 = temp2 * temp2;
population[i].fitness = x1+x2;
}
}
//计算目标值
//目标函数为f(x) = x1* x1 + x2*x2
void calculateObjectValue() {
int i;
long temp1, temp2;
double x1, x2;
for (i = 0; i < PopSize; i++) {
temp1 = decomdeChromosome(population[i].chrom, 0, LENGHT1);
temp2 = decomdeChromosome(population[i].chrom, LENGHT1, LENGHT2);
x1 = temp1 * temp1;
x2 = temp2 * temp2;
population[i].value = x1 + x2;
}
}
//把二进制转化为十进制
long decomdeChromosome(char* string, int point, int length) {
int i;
long decimal = 0L;
char * pointer;
for(i = 0, pointer=string+point; i < length;i++,pointer++){
decimal += (*pointer - '0') << (length - 1 - i);
}
return decimal;
}
//进经同时把最坏个体用目前最好个人替代///
void performEvolution() {
if (bestIndividual.fitness > currentBest.fitness) {
currentBest = population[bestIndex];
} else {
population[worstIndex] = currentBest;
}
}
//打印当前样本信息///
void outputTextReport() {
int i;
double sum;
double average;
sum = 0.0;
for (i = 0; i < PopSize; i++) {
sum += population[i].value;
}
average = sum / PopSize;
printf("gen=%d, avg=%f, best=%f",generation, average,currentBest.value);
printf(" chromosome=");
for( i = 0; i < CHROMLENGTH; i++){
printf("%c", currentBest.chrom[i]);
}
printf("/n");
}
输出结果:
[c-sharp]
view plaincopy
gen=1, avg=51.400000, best=74.000000 chromosome=101111
gen=2, avg=58.300000, best=74.000000 chromosome=101111
gen=3, avg=66.300000, best=74.000000 chromosome=101111
gen=4, avg=71.400000, best=74.000000 chromosome=101111
gen=5, avg=75.100000, best=98.000000 chromosome=111111
gen=6, avg=83.600000, best=98.000000 chromosome=111111
gen=7, avg=88.400000, best=98.000000 chromosome=111111
gen=8, avg=90.800000, best=98.000000 chromosome=111111
gen=9, avg=93.200000, best=98.000000 chromosome=111111
gen=10, avg=98.000000, best=98.000000 chromosome=111111
gen=11, avg=98.000000, best=98.000000 chromosome=111111
gen=12, avg=98.000000, best=98.000000 chromosome=111111
gen=13, avg=98.000000, best=98.000000 chromosome=111111
gen=14, avg=98.000000, best=98.000000 chromosome=111111
gen=15, avg=98.000000, best=98.000000 chromosome=111111
gen=16, avg=98.000000, best=98.000000 chromosome=111111
gen=17, avg=98.000000, best=98.000000 chromosome=111111
gen=18, avg=98.000000, best=98.000000 chromosome=111111
gen=19, avg=98.000000, best=98.000000 chromosome=111111
gen=20, avg=98.000000, best=98.000000 chromosome=111111
gen=21, avg=98.000000, best=98.000000 chromosome=111111
gen=22, avg=98.000000, best=98.000000 chromosome=111111
gen=23, avg=98.000000, best=98.000000 chromosome=111111
gen=24, avg=98.000000, best=98.000000 chromosome=111111
gen=25, avg=98.000000, best=98.000000 chromosome=111111
gen=26, avg=98.000000, best=98.000000 chromosome=111111
gen=27, avg=98.000000, best=98.000000 chromosome=111111
gen=28, avg=98.000000, best=98.000000 chromosome=111111
gen=29, avg=98.000000, best=98.000000 chromosome=111111
gen=30, avg=98.000000, best=98.000000 chromosome=111111
gen=31, avg=98.000000, best=98.000000 chromosome=111111
gen=32, avg=98.000000, best=98.000000 chromosome=111111
gen=33, avg=98.000000, best=98.000000 chromosome=111111
gen=34, avg=98.000000, best=98.000000 chromosome=111111
gen=35, avg=98.000000, best=98.000000 chromosome=111111
gen=36, avg=98.000000, best=98.000000 chromosome=111111
gen=37, avg=98.000000, best=98.000000 chromosome=111111
gen=38, avg=98.000000, best=98.000000 chromosome=111111
gen=39, avg=98.000000, best=98.000000 chromosome=111111
gen=40, avg=98.000000, best=98.000000 chromosome=111111
gen=41, avg=98.000000, best=98.000000 chromosome=111111
gen=42, avg=98.000000, best=98.000000 chromosome=111111
gen=43, avg=98.000000, best=98.000000 chromosome=111111
gen=44, avg=98.000000, best=98.000000 chromosome=111111
gen=45, avg=98.000000, best=98.000000 chromosome=111111
gen=46, avg=98.000000, best=98.000000 chromosome=111111
gen=47, avg=98.000000, best=98.000000 chromosome=111111
gen=48, avg=98.000000, best=98.000000 chromosome=111111
gen=49, avg=98.000000, best=98.000000 chromosome=111111
gen=50, avg=98.000000, best=98.000000 chromosome=111111
gen=51, avg=98.000000, best=98.000000 chromosome=111111
gen=52, avg=98.000000, best=98.000000 chromosome=111111
gen=53, avg=98.000000, best=98.000000 chromosome=111111
gen=54, avg=98.000000, best=98.000000 chromosome=111111
gen=55, avg=98.000000, best=98.000000 chromosome=111111
gen=56, avg=98.000000, best=98.000000 chromosome=111111
gen=57, avg=98.000000, best=98.000000 chromosome=111111
gen=58, avg=98.000000, best=98.000000 chromosome=111111
gen=59, avg=98.000000, best=98.000000 chromosome=111111
gen=60, avg=98.000000, best=98.000000 chromosome=111111
gen=61, avg=98.000000, best=98.000000 chromosome=111111
gen=62, avg=98.000000, best=98.000000 chromosome=111111
gen=63, avg=98.000000, best=98.000000 chromosome=111111
gen=64, avg=98.000000, best=98.000000 chromosome=111111
gen=65, avg=98.000000, best=98.000000 chromosome=111111
gen=66, avg=98.000000, best=98.000000 chromosome=111111
gen=67, avg=98.000000, best=98.000000 chromosome=111111
gen=68, avg=98.000000, best=98.000000 chromosome=111111
gen=69, avg=98.000000, best=98.000000 chromosome=111111
gen=70, avg=98.000000, best=98.000000 chromosome=111111
gen=71, avg=98.000000, best=98.000000 chromosome=111111
gen=72, avg=98.000000, best=98.000000 chromosome=111111
gen=73, avg=98.000000, best=98.000000 chromosome=111111
gen=74, avg=98.000000, best=98.000000 chromosome=111111
gen=75, avg=98.000000, best=98.000000 chromosome=111111
gen=76, avg=98.000000, best=98.000000 chromosome=111111
gen=77, avg=98.000000, best=98.000000 chromosome=111111
gen=78, avg=98.000000, best=98.000000 chromosome=111111
gen=79, avg=98.000000, best=98.000000 chromosome=111111
gen=80, avg=98.000000, best=98.000000 chromosome=111111
gen=81, avg=98.000000, best=98.000000 chromosome=111111
gen=82, avg=98.000000, best=98.000000 chromosome=111111
gen=83, avg=98.000000, best=98.000000 chromosome=111111
gen=84, avg=98.000000, best=98.000000 chromosome=111111
gen=85, avg=98.000000, best=98.000000 chromosome=111111
gen=86, avg=98.000000, best=98.000000 chromosome=111111
gen=87, avg=98.000000, best=98.000000 chromosome=111111
gen=88, avg=98.000000, best=98.000000 chromosome=111111
gen=89, avg=98.000000, best=98.000000 chromosome=111111
gen=90, avg=98.000000, best=98.000000 chromosome=111111
gen=91, avg=98.000000, best=98.000000 chromosome=111111
gen=92, avg=98.000000, best=98.000000 chromosome=111111
gen=93, avg=98.000000, best=98.000000 chromosome=111111
gen=94, avg=98.000000, best=98.000000 chromosome=111111
gen=95, avg=98.000000, best=98.000000 chromosome=111111
gen=96, avg=98.000000, best=98.000000 chromosome=111111
gen=97, avg=98.000000, best=98.000000 chromosome=111111
gen=98, avg=98.000000, best=98.000000 chromosome=111111
gen=99, avg=98.000000, best=98.000000 chromosome=111111
gen=100, avg=98.000000, best=98.000000 chromosome=111111
请按任意键继续. . .
从结果可以看出,遗传算法收敛得非常快,在第5代的时候,已经达到了全局最优解.如果把初始种群扩大,收敛得会更快.
遗传算法主要应用领域:
(1)组合优化 (2)函数优化
(3)自动控制 (4)生产调度
(5)图像处理 (6)机器学习
(7)人工生命 (8)数据挖掘
为更好地理解遗传算法的运算过程,下面用手工计算来简单地模拟遗传算法的各
个主要执行步骤。
例:求下述二元函数的最大值:
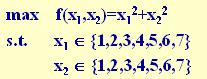
(1) 个体编码
遗传算法的运算对象是表示个体的符号串,所以必须把变量 x1, x2 编码为一种
符号串。本题中,用无符号二进制整数来表示。
因 x1, x2 为 0 ~ 7之间的整数,所以分别用3位无符号二进制整数来表示,将它
们连接在一起所组成的6位无符号二进制数就形成了个体的基因型,表示一个可
行解。
例如,基因型 X=101110 所对应的表现型是:x=[ 5,6 ]。
个体的表现型x和基因型X之间可通过编码和解码程序相互转换。
(2) 初始群体的产生
遗传算法是对群体进行的进化操作,需要给其淮备一些表示起始搜索点的初始
群体数据。
本例中,群体规模的大小取为4,即群体由4个个体组成,每个个体可通过随机
方法产生。
如:011101,101011,011100,111001
(3) 适应度汁算
遗传算法中以个体适应度的大小来评定各个个体的优劣程度,从而决定其遗传
机会的大小。
本例中,目标函数总取非负值,并且是以求函数最大值为优化目标,故可直接
利用目标函数值作为个体的适应度。
(4) 选择运算
选择运算(或称为复制运算)把当前群体中适应度较高的个体按某种规则或模型遗传到下一代群体中。一般要求适应度较高的个体将有更多的机会遗传到下一代
群体中。
本例中,我们采用与适应度成正比的概率来确定各个个体复制到下一代群体中
的数量。其具体操作过程是:
• 先计算出群体中所有个体的适应度的总和 fi ( i=1.2,…,M );
• 其次计算出每个个体的相对适应度的大小 fi / fi ,它即为每个个体被遗传
到下一代群体中的概率,
• 每个概率值组成一个区域,全部概率值之和为1;
• 最后再产生一个0到1之间的随机数,依据该随机数出现在上述哪一个概率区
域内来确定各个个体被选中的次数。

(5) 交叉运算
交叉运算是遗传算法中产生新个体的主要操作过程,它以某一概率相互交换某
两个个体之间的部分染色体。
本例采用单点交叉的方法,其具体操作过程是:
• 先对群体进行随机配对;
• 其次随机设置交叉点位置;
• 最后再相互交换配对染色体之间的部分基因。

(6) 变异运算
变异运算是对个体的某一个或某一些基因座上的基因值按某一较小的概率进
行改变,它也是产生新个体的一种操作方法。
本例中,我们采用基本位变异的方法来进行变异运算,其具体操作过程是:
• 首先确定出各个个体的基因变异位置,下表所示为随机产生的变异点位置,
其中的数字表示变异点设置在该基因座处;
• 然后依照某一概率将变异点的原有基因值取反。

对群体P(t)进行一轮选择、交叉、变异运算之后可得到新一代的群体p(t+1)。

从上表中可以看出,群体经过一代进化之后,其适应度的最大值、平均值都得
到了明显的改进。事实上,这里已经找到了最佳个体“111111”。
[注意]
需要说明的是,表中有些栏的数据是随机产生的。这里为了更好地说明问题,
我们特意选择了一些较好的数值以便能够得到较好的结果,而在实际运算过程中
有可能需要一定的循环次数才能达到这个最优结果。

四.C语言实现
#include <stdio.h>
#include <stdlib.h>
#include <time.h>
/////////////The definiton of user data////
#define Cmax 100 //certain maximal value
#define Cmin 0 //certain minimum value
#define LENGHT1 3 //the chromosome length of 1st variable
#define LENGHT2 3 //the chromosome length of 2nd variable
//总染体长度
#define CHROMLENGTH LENGHT1+LENGHT2
const int MaxGeneration = 100; //最大代数
const int PopSize = 10; //样本大小
const double Pc = 0.6; //交叉概率
const double Pm = 0.001; //变异概率
////////////// 数据结构定义///////////////////
struct Individual{
char chrom[CHROMLENGTH + 1]; //一个个体的染色体
double value; //目标值
double fitness; //适应度
};
int generation ; //进化次数
int bestIndex; //最好个体的下标
int worstIndex; //最坏个体的游标
Individual bestIndividual ; //当前一代中的最好个体
Individual worstIndividual ; ///当前一代中的坏个体
// best individual by now
Individual currentBest ;// 到目前为止的最好个体
Individual population [PopSize] ;//样本
///////////////////////
void generateInitialPopulation();
void generateNextPopulation();
void evalutePopulation();
long decomdeChromosome(char*, int, int);
void calculateObjectValue();
void calculateFitnessValue();
void findBestAndWorstIndividual();
void performEvolution();
void selectionOperator();
void
109bf
crossoverOperator();
void mutationOperator();
void outputTextReport();
//////////////////////////////////////////////
int main(){
generation = 0;
generateInitialPopulation();
evalutePopulation();
while (generation < MaxGeneration) {
generation++;
generateNextPopulation();
evalutePopulation();
performEvolution();
outputTextReport();
}
return 0;
}
//////////////////////////////////////////////////////////////
//////产生第一代样本/////
void generateInitialPopulation() {
int i, j;
srand((unsigned)time(NULL));
for (i = 0; i < PopSize; i++) {
for (j = 0; j < CHROMLENGTH; j++) {
population[i].chrom[j] = ((rand() % 10) < 5) ? '0' : '1';
}
population[i].chrom[CHROMLENGTH] = '/0';
}
}
////////产生下一代样本 //////
void generateNextPopulation() {
selectionOperator();
crossoverOperator();
mutationOperator();
}
//变异算子//
void mutationOperator() {
int i, j;
double p;
// bit mutation
for (i = 0; i < PopSize; i++) {
for (j = 0; j < CHROMLENGTH; j++) {
p = rand() % 1000 / 1000.0;
if (p < Pm) {
population[i].chrom[j] = (population[i].chrom[j] == '0') ? '1': '0';
}
}
}
}
//交叉算子///
void crossoverOperator() {
int i, j;
int index[PopSize];
int point, temp;
double p;
char ch;
for (i = 0; i < PopSize; i++) {
index[i] = i;
}
for (i = 0; i < PopSize; i++) {
point = rand() %(PopSize - i);
temp = index[i];
index[i] = index[point + i];
index[point + i] = temp;
}
for (i = 0; i < PopSize - 1; i+=2) {
p = rand() % 1000 / 1000.0;
if (p < Pc) {
point = rand()% (CHROMLENGTH - 1) + 1;
for (j = point; j < CHROMLENGTH; j++) {
ch = population[index[i]].chrom[j];
population[index[i]].chrom[j] = population[index[i + 1]].chrom[j];
population[index[i + 1]].chrom[j] = ch;
}
}
}
}
///选择算子/////////////
void selectionOperator() {
int i, index;
double p, sum = 0.0;
double cfitness[PopSize];
Individual newpopulation[PopSize];
for (i = 0; i < PopSize; i++) {
sum += population[i].fitness;
}
for (i = 0; i < PopSize; i++) {
cfitness[i] = population[i].fitness / sum;
}
// calculate cumulative fitness
for (i = 1; i < PopSize; i++) {
cfitness[i] = cfitness[i] + cfitness[i - 1];
}
for (i = 0; i < PopSize; i++) {
p = rand() % 1000 / 1000.0;
index = 0;
while (p > cfitness[index]) {
index++;
}
newpopulation[i] = population[index];
}
for (i = 0; i < PopSize; i++) {
population[i] = newpopulation[i];
}
}
/////依据某些公式对样本进行评价////
void evalutePopulation() {
calculateObjectValue();
calculateFitnessValue();
findBestAndWorstIndividual();
}
//找出到目前为止最好的个体//////
void findBestAndWorstIndividual() {
int i;
double sum = 0.0;
bestIndividual = population[0];
worstIndividual = population[0];
for (i = 0; i < PopSize; i++) {
if (population[i].fitness > bestIndividual.fitness) {
bestIndividual = population[i];
bestIndex = i;
} else if (population[i].fitness < worstIndividual.fitness) {
worstIndividual = population[i];
worstIndex = i;
}
sum += population[i].fitness;
}
if (generation == 0) {
currentBest = bestIndividual;
} else {
if (bestIndividual.fitness > currentBest.fitness) {
currentBest = bestIndividual;
}
}
}
//计算适应度///
void calculateFitnessValue() {
int i;
long temp1, temp2;
double x1, x2;
for (i = 0; i < PopSize; i++) {
temp1 = decomdeChromosome(population[i].chrom, 0, LENGHT1);
temp2 = decomdeChromosome(population[i].chrom, LENGHT1, LENGHT2);
x1 = temp1 * temp1;
x2 = temp2 * temp2;
population[i].fitness = x1+x2;
}
}
//计算目标值
//目标函数为f(x) = x1* x1 + x2*x2
void calculateObjectValue() {
int i;
long temp1, temp2;
double x1, x2;
for (i = 0; i < PopSize; i++) {
temp1 = decomdeChromosome(population[i].chrom, 0, LENGHT1);
temp2 = decomdeChromosome(population[i].chrom, LENGHT1, LENGHT2);
x1 = temp1 * temp1;
x2 = temp2 * temp2;
population[i].value = x1 + x2;
}
}
//把二进制转化为十进制
long decomdeChromosome(char* string, int point, int length) {
int i;
long decimal = 0L;
char * pointer;
for(i = 0, pointer=string+point; i < length;i++,pointer++){
decimal += (*pointer - '0') << (length - 1 - i);
}
return decimal;
}
//进经同时把最坏个体用目前最好个人替代///
void performEvolution() {
if (bestIndividual.fitness > currentBest.fitness) {
currentBest = population[bestIndex];
} else {
population[worstIndex] = currentBest;
}
}
//打印当前样本信息///
void outputTextReport() {
int i;
double sum;
double average;
sum = 0.0;
for (i = 0; i < PopSize; i++) {
sum += population[i].value;
}
average = sum / PopSize;
printf("gen=%d, avg=%f, best=%f",generation, average,currentBest.value);
printf(" chromosome=");
for( i = 0; i < CHROMLENGTH; i++){
printf("%c", currentBest.chrom[i]);
}
printf("/n");
}
输出结果:
[c-sharp]
view plaincopy
gen=1, avg=51.400000, best=74.000000 chromosome=101111
gen=2, avg=58.300000, best=74.000000 chromosome=101111
gen=3, avg=66.300000, best=74.000000 chromosome=101111
gen=4, avg=71.400000, best=74.000000 chromosome=101111
gen=5, avg=75.100000, best=98.000000 chromosome=111111
gen=6, avg=83.600000, best=98.000000 chromosome=111111
gen=7, avg=88.400000, best=98.000000 chromosome=111111
gen=8, avg=90.800000, best=98.000000 chromosome=111111
gen=9, avg=93.200000, best=98.000000 chromosome=111111
gen=10, avg=98.000000, best=98.000000 chromosome=111111
gen=11, avg=98.000000, best=98.000000 chromosome=111111
gen=12, avg=98.000000, best=98.000000 chromosome=111111
gen=13, avg=98.000000, best=98.000000 chromosome=111111
gen=14, avg=98.000000, best=98.000000 chromosome=111111
gen=15, avg=98.000000, best=98.000000 chromosome=111111
gen=16, avg=98.000000, best=98.000000 chromosome=111111
gen=17, avg=98.000000, best=98.000000 chromosome=111111
gen=18, avg=98.000000, best=98.000000 chromosome=111111
gen=19, avg=98.000000, best=98.000000 chromosome=111111
gen=20, avg=98.000000, best=98.000000 chromosome=111111
gen=21, avg=98.000000, best=98.000000 chromosome=111111
gen=22, avg=98.000000, best=98.000000 chromosome=111111
gen=23, avg=98.000000, best=98.000000 chromosome=111111
gen=24, avg=98.000000, best=98.000000 chromosome=111111
gen=25, avg=98.000000, best=98.000000 chromosome=111111
gen=26, avg=98.000000, best=98.000000 chromosome=111111
gen=27, avg=98.000000, best=98.000000 chromosome=111111
gen=28, avg=98.000000, best=98.000000 chromosome=111111
gen=29, avg=98.000000, best=98.000000 chromosome=111111
gen=30, avg=98.000000, best=98.000000 chromosome=111111
gen=31, avg=98.000000, best=98.000000 chromosome=111111
gen=32, avg=98.000000, best=98.000000 chromosome=111111
gen=33, avg=98.000000, best=98.000000 chromosome=111111
gen=34, avg=98.000000, best=98.000000 chromosome=111111
gen=35, avg=98.000000, best=98.000000 chromosome=111111
gen=36, avg=98.000000, best=98.000000 chromosome=111111
gen=37, avg=98.000000, best=98.000000 chromosome=111111
gen=38, avg=98.000000, best=98.000000 chromosome=111111
gen=39, avg=98.000000, best=98.000000 chromosome=111111
gen=40, avg=98.000000, best=98.000000 chromosome=111111
gen=41, avg=98.000000, best=98.000000 chromosome=111111
gen=42, avg=98.000000, best=98.000000 chromosome=111111
gen=43, avg=98.000000, best=98.000000 chromosome=111111
gen=44, avg=98.000000, best=98.000000 chromosome=111111
gen=45, avg=98.000000, best=98.000000 chromosome=111111
gen=46, avg=98.000000, best=98.000000 chromosome=111111
gen=47, avg=98.000000, best=98.000000 chromosome=111111
gen=48, avg=98.000000, best=98.000000 chromosome=111111
gen=49, avg=98.000000, best=98.000000 chromosome=111111
gen=50, avg=98.000000, best=98.000000 chromosome=111111
gen=51, avg=98.000000, best=98.000000 chromosome=111111
gen=52, avg=98.000000, best=98.000000 chromosome=111111
gen=53, avg=98.000000, best=98.000000 chromosome=111111
gen=54, avg=98.000000, best=98.000000 chromosome=111111
gen=55, avg=98.000000, best=98.000000 chromosome=111111
gen=56, avg=98.000000, best=98.000000 chromosome=111111
gen=57, avg=98.000000, best=98.000000 chromosome=111111
gen=58, avg=98.000000, best=98.000000 chromosome=111111
gen=59, avg=98.000000, best=98.000000 chromosome=111111
gen=60, avg=98.000000, best=98.000000 chromosome=111111
gen=61, avg=98.000000, best=98.000000 chromosome=111111
gen=62, avg=98.000000, best=98.000000 chromosome=111111
gen=63, avg=98.000000, best=98.000000 chromosome=111111
gen=64, avg=98.000000, best=98.000000 chromosome=111111
gen=65, avg=98.000000, best=98.000000 chromosome=111111
gen=66, avg=98.000000, best=98.000000 chromosome=111111
gen=67, avg=98.000000, best=98.000000 chromosome=111111
gen=68, avg=98.000000, best=98.000000 chromosome=111111
gen=69, avg=98.000000, best=98.000000 chromosome=111111
gen=70, avg=98.000000, best=98.000000 chromosome=111111
gen=71, avg=98.000000, best=98.000000 chromosome=111111
gen=72, avg=98.000000, best=98.000000 chromosome=111111
gen=73, avg=98.000000, best=98.000000 chromosome=111111
gen=74, avg=98.000000, best=98.000000 chromosome=111111
gen=75, avg=98.000000, best=98.000000 chromosome=111111
gen=76, avg=98.000000, best=98.000000 chromosome=111111
gen=77, avg=98.000000, best=98.000000 chromosome=111111
gen=78, avg=98.000000, best=98.000000 chromosome=111111
gen=79, avg=98.000000, best=98.000000 chromosome=111111
gen=80, avg=98.000000, best=98.000000 chromosome=111111
gen=81, avg=98.000000, best=98.000000 chromosome=111111
gen=82, avg=98.000000, best=98.000000 chromosome=111111
gen=83, avg=98.000000, best=98.000000 chromosome=111111
gen=84, avg=98.000000, best=98.000000 chromosome=111111
gen=85, avg=98.000000, best=98.000000 chromosome=111111
gen=86, avg=98.000000, best=98.000000 chromosome=111111
gen=87, avg=98.000000, best=98.000000 chromosome=111111
gen=88, avg=98.000000, best=98.000000 chromosome=111111
gen=89, avg=98.000000, best=98.000000 chromosome=111111
gen=90, avg=98.000000, best=98.000000 chromosome=111111
gen=91, avg=98.000000, best=98.000000 chromosome=111111
gen=92, avg=98.000000, best=98.000000 chromosome=111111
gen=93, avg=98.000000, best=98.000000 chromosome=111111
gen=94, avg=98.000000, best=98.000000 chromosome=111111
gen=95, avg=98.000000, best=98.000000 chromosome=111111
gen=96, avg=98.000000, best=98.000000 chromosome=111111
gen=97, avg=98.000000, best=98.000000 chromosome=111111
gen=98, avg=98.000000, best=98.000000 chromosome=111111
gen=99, avg=98.000000, best=98.000000 chromosome=111111
gen=100, avg=98.000000, best=98.000000 chromosome=111111
请按任意键继续. . .
从结果可以看出,遗传算法收敛得非常快,在第5代的时候,已经达到了全局最优解.如果把初始种群扩大,收敛得会更快.
遗传算法主要应用领域:
(1)组合优化 (2)函数优化
(3)自动控制 (4)生产调度
(5)图像处理 (6)机器学习
(7)人工生命 (8)数据挖掘
相关文章推荐
- 遗传算法入门例子和总结
- 自定义标签入门例子流程总结
- 遗传算法入门例子和总结
- Hibernate入门小例子的总结
- Hibernate入门之自己写的小例子的总结
- Ajax 入门例子总结
- 一个非常好的理解遗传算法的例子 强烈推荐入门
- Hibernate入门之自己写的小例子的总结 zz
- 遗传算法入门程序例子
- Shiro学习总结(二)--Shiro的入门小例子
- [总结]如何利用反射得到中间层中的所有类(利用XPO来做的项目中的例子)
- [总结]发送邮件共有几种方法,测试成功的例子
- 一个经典的ADO.NET入门例子
- 遗传算法入门(连载之六)
- 遗传算法入门(连载之十) 神经网络入门(连载预告)
- VB Api简单入门(2)-一个简单的例子
- 遗传算法入门(连载之七)
- [总结]客户端JavaScript读取Xml文档的例子!
- 遗传算法入门(连载之二)
- 遗传算法入门(连载之四)
