TCGA数据下载:R包TCGA2STAT介绍
2016-10-13 23:18
429 查看
上期介绍了若干种获取TCGA数据的方法,今天这期会落点于TCGA2STAT这个R包的介绍上,一步步的来说明下载方法,哪些数据是可以下载到的。
R包的下载
选择如何的镜像,咱们在中国,就选择china,这样的话下载速度会很快,也容易安装R包成功。
可下载的数据
1、 RNASeq ,默认是count类型,是指下载raw read counts数据。可以改为RPKM,是指下载normalized read counts数据 (reads per
kilobase per million mapped reads)。
2、RNASeq2,是指来自the second pipeline的RNASeq基因数据。
3、miRNASeq,默认为count,是指下载raw read counts数据;可以改为rpmmm,是指下载normalized read counts。
4、Mutation,默认为smoatic,是指non-silent somatic mutations 数据;改为all,表示为all mutations数据。
5、Methylation,默认为来自platform为27K;platform可改为450K。
6、CNA_CGH,默认为415K,是指CGH Custom Microarray 2x415K ;可改为244A,是指 CGH Microarray。
7、mRNA_Array,默认为G450,是指Agilent 244K Custom Gene Expression G4502A ;可改为U133,只指Affymetrix Human Genome U133A 2.0 Array;还可以改为Huex,是指Affymetrix Human Exon 1.0。
下载格式
上面的参数的值都为默认情况下的,disease包括了33种,”ACC”, “BLCA”, “BRCA”, “CESC”, “CHOL”, “COAD”, “COADREAD”, “DLBC”,”ESCA”, “FPPP”, “GBM”, “GBMLGG”, “HNSC”, “KICH”, “KIPAN”, “KIRC”, “KIRP”, “LAML”, “LGG”,”LIHC”, “LUAD”, “LUSC”, “MESO”, “OV”, “PAAD”, “PCPG”, “PRAD”, “READ”, “SARC”, “SKCM”,”STAD”, “TGCT”, “THCA”, “THYM”, “UCEC”, “UCS”, and “UVM”。
根据TCGA官网给出的图,介绍了目前收集到的数据情况:
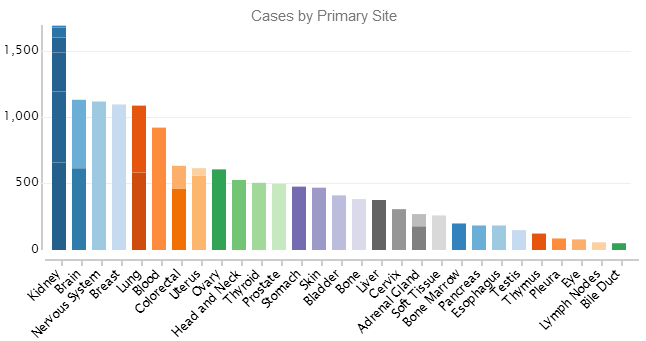
纵轴表示收集到的病例数。
下面来举一个例子来说明数的下载:
如果在win系统下,会报错:
Error: TAR is not installed in the system. Data unzip failed.
谷歌查到说需要安装Cygwin软件,然后通过R语言命令来加载,而且每次使用都必须做加载,命令如下:
个人见解
TCGA2STAT这个包下载数据效果太差,不稳定,时常没法下载完就中断了,及其不稳定。也许你所在地方刚好能很好的下载也说不定。
R包的下载
install.packages("TCGA2STAT")选择如何的镜像,咱们在中国,就选择china,这样的话下载速度会很快,也容易安装R包成功。
可下载的数据
1、 RNASeq ,默认是count类型,是指下载raw read counts数据。可以改为RPKM,是指下载normalized read counts数据 (reads per
kilobase per million mapped reads)。
2、RNASeq2,是指来自the second pipeline的RNASeq基因数据。
3、miRNASeq,默认为count,是指下载raw read counts数据;可以改为rpmmm,是指下载normalized read counts。
4、Mutation,默认为smoatic,是指non-silent somatic mutations 数据;改为all,表示为all mutations数据。
5、Methylation,默认为来自platform为27K;platform可改为450K。
6、CNA_CGH,默认为415K,是指CGH Custom Microarray 2x415K ;可改为244A,是指 CGH Microarray。
7、mRNA_Array,默认为G450,是指Agilent 244K Custom Gene Expression G4502A ;可改为U133,只指Affymetrix Human Genome U133A 2.0 Array;还可以改为Huex,是指Affymetrix Human Exon 1.0。
下载格式
getTCGA(disease = "GBM", data.type = "RNASeq2", type = "", filter = "Y",
p = getOption("mc.cores", 2L), clinical = FALSE, cvars = "OS")上面的参数的值都为默认情况下的,disease包括了33种,”ACC”, “BLCA”, “BRCA”, “CESC”, “CHOL”, “COAD”, “COADREAD”, “DLBC”,”ESCA”, “FPPP”, “GBM”, “GBMLGG”, “HNSC”, “KICH”, “KIPAN”, “KIRC”, “KIRP”, “LAML”, “LGG”,”LIHC”, “LUAD”, “LUSC”, “MESO”, “OV”, “PAAD”, “PCPG”, “PRAD”, “READ”, “SARC”, “SKCM”,”STAD”, “TGCT”, “THCA”, “THYM”, “UCEC”, “UCS”, and “UVM”。
根据TCGA官网给出的图,介绍了目前收集到的数据情况:

纵轴表示收集到的病例数。
下面来举一个例子来说明数的下载:
library(TCGA2STAT) BRCA <- getTCGA(disease = "BRCA", data.type = "RNASeq",type = "count", clinical=TRUE)
如果在win系统下,会报错:
Error: TAR is not installed in the system. Data unzip failed.
谷歌查到说需要安装Cygwin软件,然后通过R语言命令来加载,而且每次使用都必须做加载,命令如下:
Sys.setenv(TAR="D:/cygwin64/bin/tar",R_GZIPCMD="D:/cygwin64/bin/gzip")
个人见解
TCGA2STAT这个包下载数据效果太差,不稳定,时常没法下载完就中断了,及其不稳定。也许你所在地方刚好能很好的下载也说不定。
相关文章推荐
- TCGA数据下载:R包TCGAbiolinks介绍
- TCGA数据下载:R包RTCGA介绍
- LANDSAT数据下载及数据格式介绍
- TCGA-Assembler工具下载数据
- 介绍一种遥感数据的下载方法
- TCGA数据下载方法简介
- TCGA数据文件下载
- LANDSAT数据下载及数据格式介绍
- Essential Grouping高性能的数据分组引擎介绍及下载
- TCGA数据下载:R包RTCGAToolbox介绍
- 详细介绍ASP.NET中WebClient方法示例下载远程数据
- TCGA数据下载和整理工具----GDCRNATools
- 列车时刻表查询器0.6版(SmartPhone版)发布,采用了SQLite数据引擎,欢迎下载试用。
- J2ME加密数据的一个第三方开源免费类库介绍
- 教程下载板块数据层完成!
- Java 数据对象(JDO)介绍(三)
- 本文介绍了一种利用Repeater控件显示主-从关系数据表的方法。
- 实例介绍文件下载,注册表操作等基本技巧!(原创)
- Java 数据对象(JDO)介绍(四)
- MySQL数据导入导出方法与工具介绍
