基于MITK实现区域生长分割的介绍
2016-06-22 20:28
246 查看
本文由Markdown语法编辑器编辑完成。
与MITK同名的还有另一款由中科院自动化研究所开发的软件,全称为”Medical Image Toolkit, MITK“。官网地址为:http://www.mitk.net/index_ch1.html 。
本文介绍的MITK是指德国研究室开发的软件。在随后的博文中再介绍中科院自动化所研发的同名软件。
MITK软件的下载地址为:http://mitk.org/wiki/Downloads 。本文所演示的软件是在Windows 7平台下的64位软件,MITK Workbench。
以下介绍在该软件上的一些基本操作:
点击工具栏中的”DICOM”字样的按钮,会弹出一个对话框,提示用户选择要导入的影像序列的位置。点击”Scan directory”可以选择本地存储影像的位置。
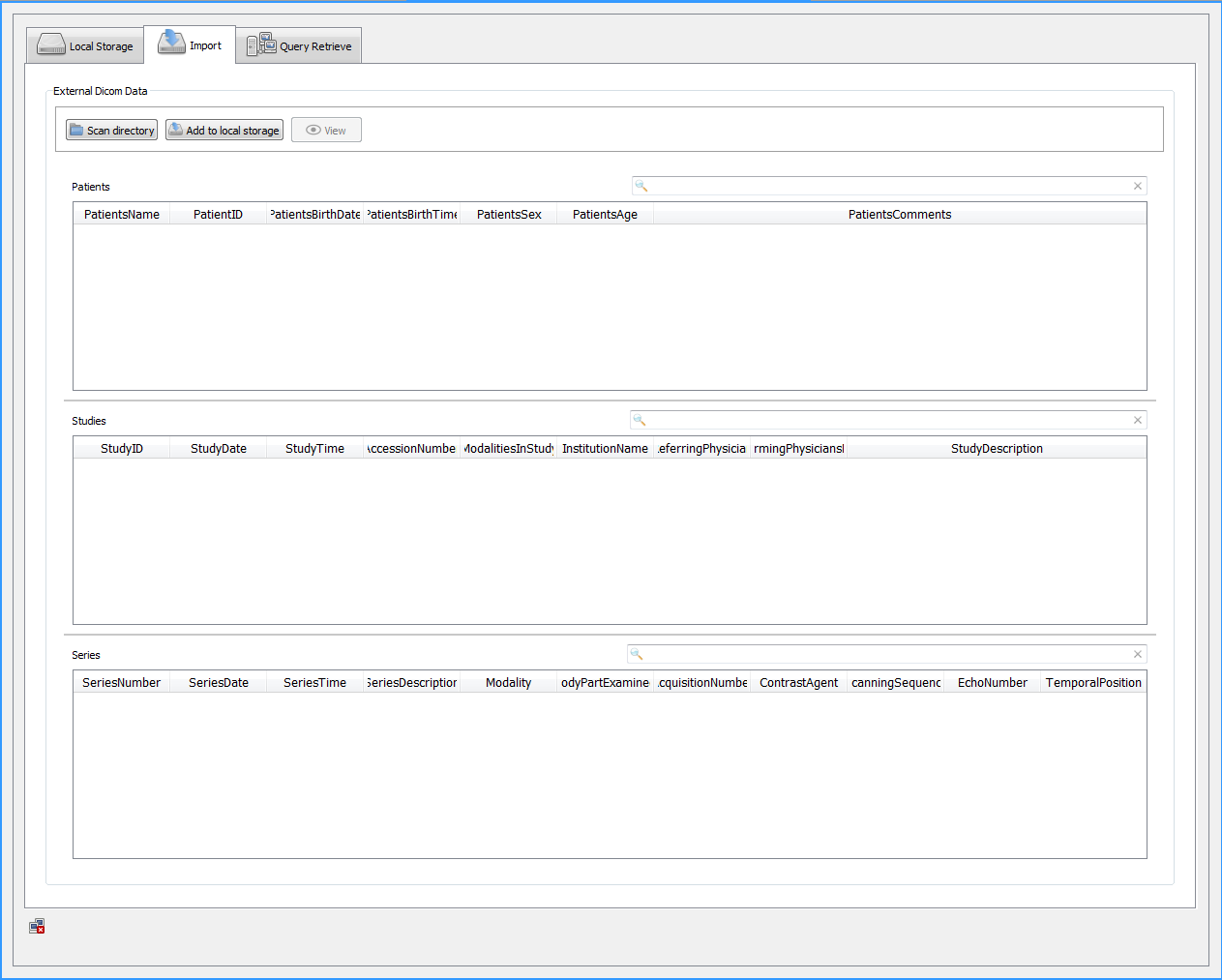
用户在本地硬盘上选择要显示的影像序列后,在MITK Dicom Browse的界面中会显示加载进来的数据,按照Patients, Studies, Series的层次进行显示。
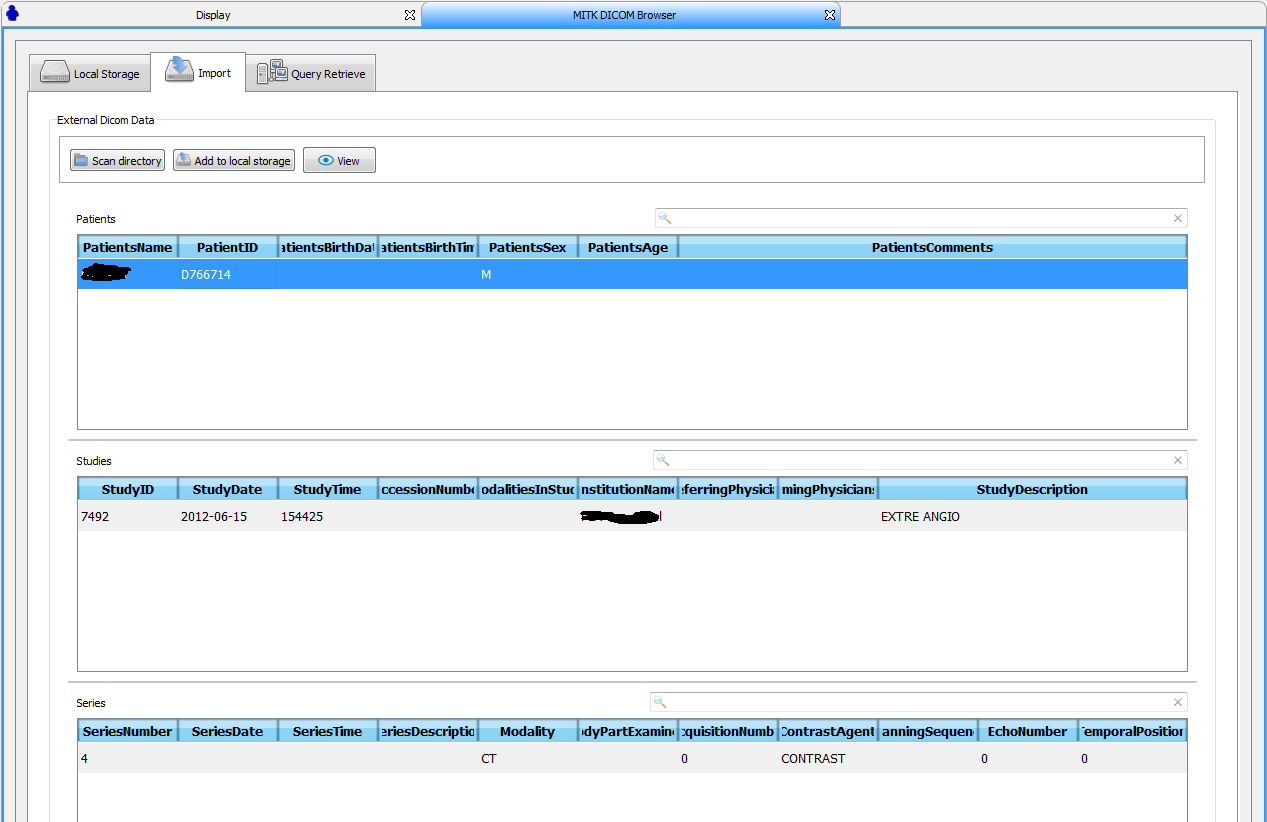
如上图所示,该患者有一个检查,这个检查中只有一个序列。
选中要显示的序列后,上方最右侧的“View”按钮变为可用状态。
这时,该序列便显示在了左侧的Data Manager方框中。
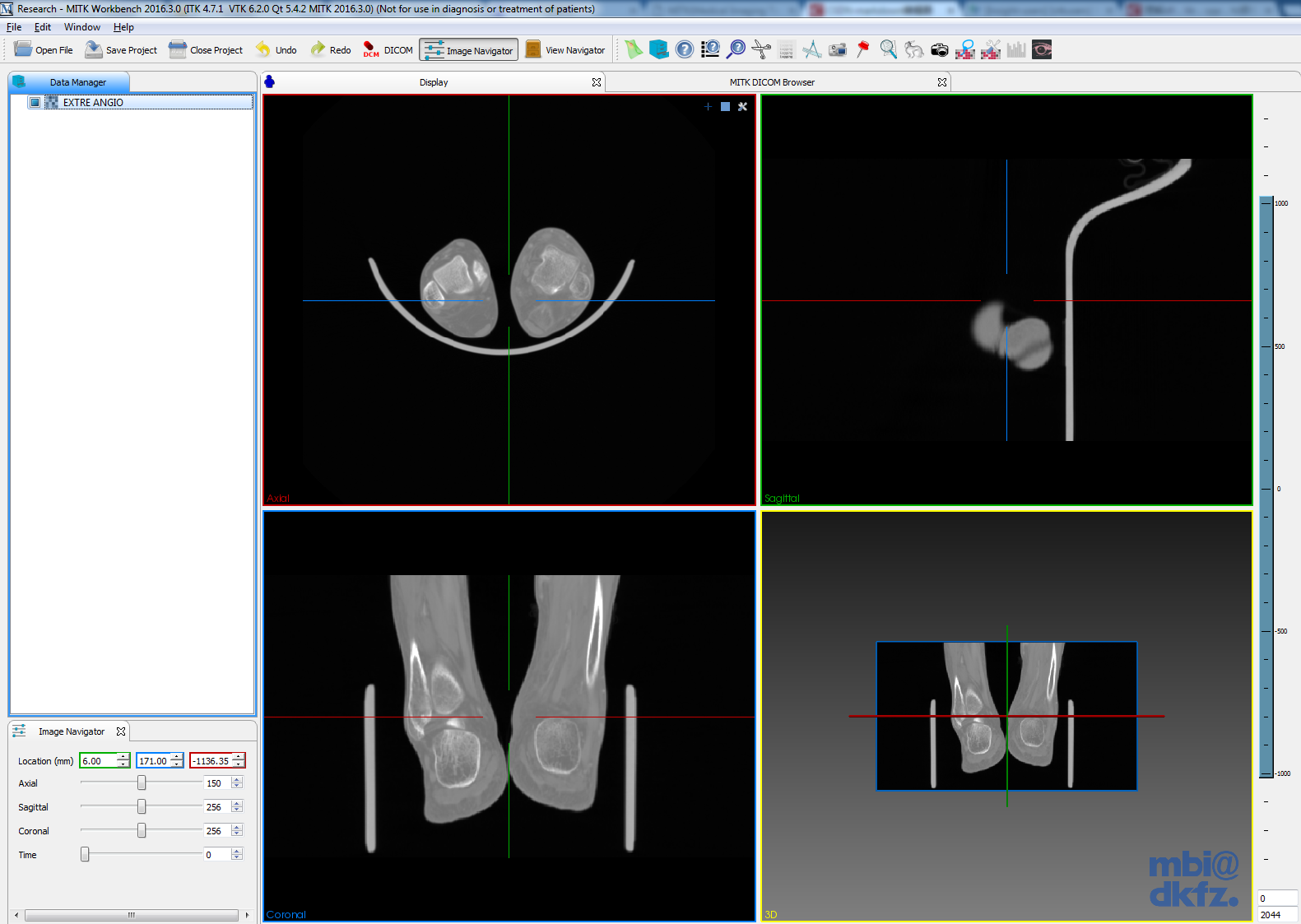
显示MPR后,在左下角的”Image Navigator”界面中,会显示MPR的轴线中心的坐标位置,以及轴、矢和冠状位三个切片的序号。
从显示的值可以看出,此时MPR轴线的中心点坐标为(6.00,171.00,-1136.35)。轴、矢、冠的序号分别为:150,256,256。
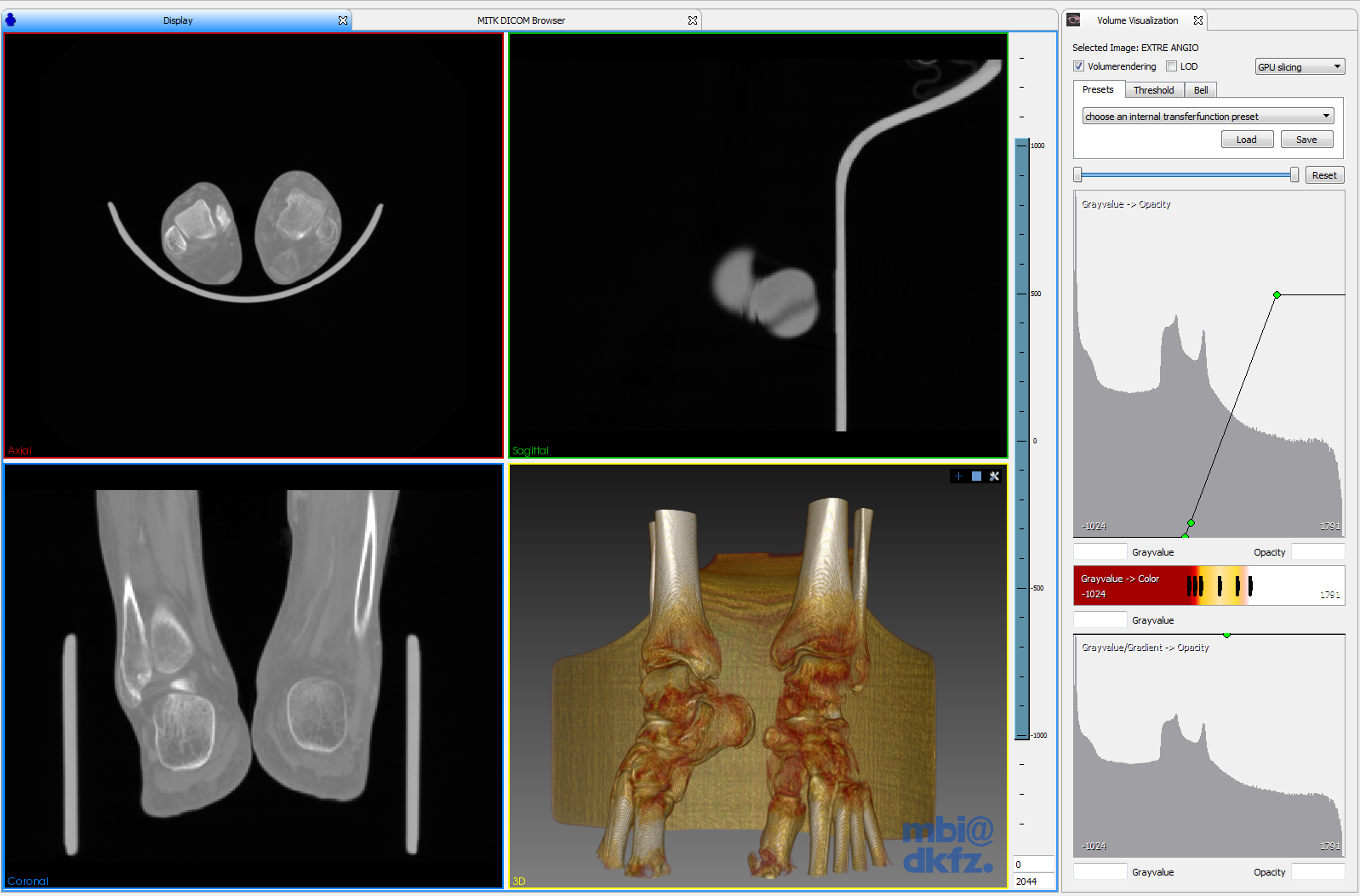
如果想要观察数据体的三维影像,需要对体数据进行Volume Rendering。
点击工具栏中的最后一个名称为Volume Visualization的按钮。此时会弹出一个界面,用来设置体渲染的一些选项。
勾选第一项Volume Rendering。则在第四个窗口中会显示数据体三维重建,并且增加了预设的颜色和透明度传递函数后的结果。
点击工具栏从右往左数第四个,名称为Segmentation的按钮,即为图像分割的按钮。
此时,会弹出一个图像分割的工具箱面板。
面板的最上面部分为Data Selection,是用户选择待分割的图像和创建保存分割结果的。
默认将体数据作为分割对象。创建一个名称为bone_segment的标签,作为保存分割结果的文件。
该分割工具箱面板提供了分割的2D Tool和3D Tool。
2D Tool包括:Add, Subtract, Correction, Paint, Wipe, Region Growing, Fill, Erase, Live Wire, 2D Fast Marching的分割功能;
3D Tool包括:Threshold, UL Threshold, Otsu, Fast Marching 3D, Region Growing 3D, Watershed, Picking的分割功能。
这里介绍Region Growing 3D算法。
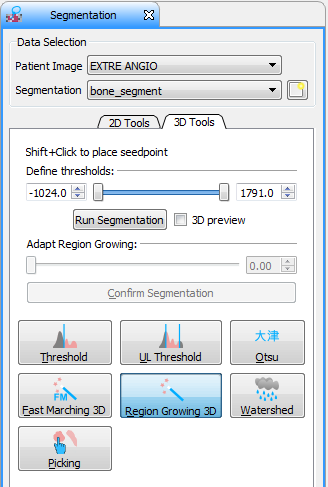
Region Growing在ITK中有不同的实现算法。通过这里的界面显示,MITK中应用的应该是ITK中的connectedThresholdImageFilter的类来实现区域生长的分割功能。
默认的阈值上下限为体数据的灰度最小值和最大值。这里载入的是CT数据,所以下限是-1024.0, 上限是1791.0。通过查阅资料,要分割的股骨区域的灰度范围约为280~1791,因此修改下限值为280。
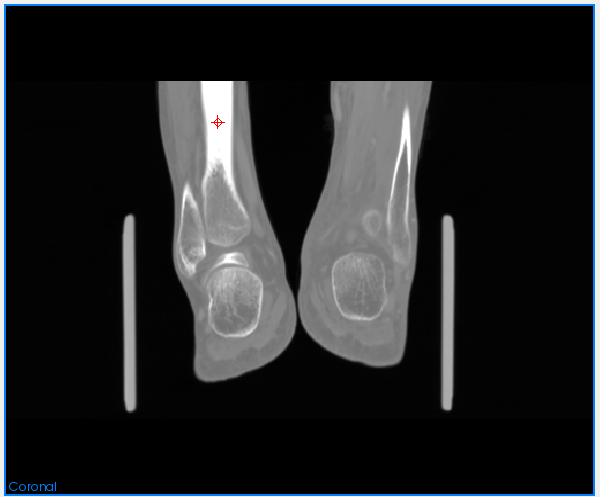
然后点击键盘上的Shift键后,同时用鼠标在MPR的任一个面上选取股骨区域的一个点作为种子点。如下图所示为,在冠状位上选取了一个种子点。
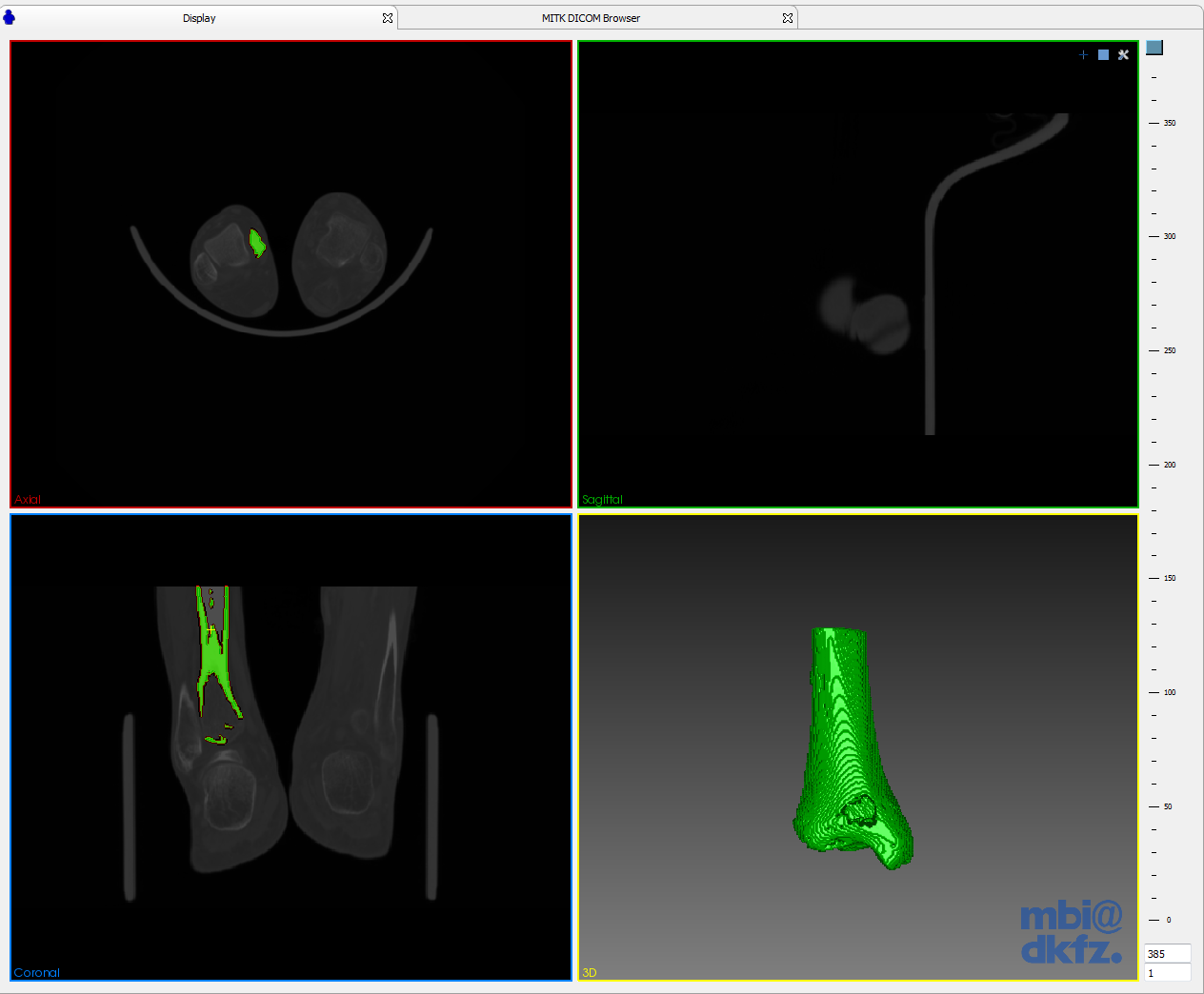
然后点击分割面板中的“Run Segmentation”按钮,即可以在MPR区域显示出区域生长后的区域。选中分割面板的3D preview选项,则会将分割后的区域进行三维重建,显示在第四个窗口中。
参考链接:
1. http://mitk.org/wiki/MITK
2. http://www.mitk.net/
3. http://www.oschina.net/p/mitk
1.关于MITK:
MITK的全称是”The Medical Imaging Interaction Toolkit”。它是一款开源的交互式医学图像处理软件开发和应用平台。MITK将ITK和VTK整合为一个应用框架。该软件由德国一个研究室开发。官网地址为:http://mitk.org/wiki/MITK 。与MITK同名的还有另一款由中科院自动化研究所开发的软件,全称为”Medical Image Toolkit, MITK“。官网地址为:http://www.mitk.net/index_ch1.html 。
本文介绍的MITK是指德国研究室开发的软件。在随后的博文中再介绍中科院自动化所研发的同名软件。
MITK软件的下载地址为:http://mitk.org/wiki/Downloads 。本文所演示的软件是在Windows 7平台下的64位软件,MITK Workbench。
2. MITK的基本操作:
以下是下载MITK的安装包,并且安装后的MITK Workbench的启动后的主界面。
以下介绍在该软件上的一些基本操作:
2.1 加载DICOM序列图像
作为一个医学影像处理软件。最基本的功能当然是导入医学影像数据了。医学影像一般是以序列的形式存放在文件夹里。点击工具栏中的”DICOM”字样的按钮,会弹出一个对话框,提示用户选择要导入的影像序列的位置。点击”Scan directory”可以选择本地存储影像的位置。

用户在本地硬盘上选择要显示的影像序列后,在MITK Dicom Browse的界面中会显示加载进来的数据,按照Patients, Studies, Series的层次进行显示。

如上图所示,该患者有一个检查,这个检查中只有一个序列。
选中要显示的序列后,上方最右侧的“View”按钮变为可用状态。
这时,该序列便显示在了左侧的Data Manager方框中。
2.2 实现MPR浏览切面的功能
在显示的序列文字上,点击鼠标右键,在右键菜单中选择第一项“Global Reinit”。这时该序列的MPR图像便会显示在右侧的三个视图中。如下图所示。
显示MPR后,在左下角的”Image Navigator”界面中,会显示MPR的轴线中心的坐标位置,以及轴、矢和冠状位三个切片的序号。
从显示的值可以看出,此时MPR轴线的中心点坐标为(6.00,171.00,-1136.35)。轴、矢、冠的序号分别为:150,256,256。
2.3 三维重建体数据的功能
除了MPR的视图外,右下角是三维视图。当加载影像并显示MPR序列时,默认会把三个层片共同显示在三维视图中。如果想要观察数据体的三维影像,需要对体数据进行Volume Rendering。
点击工具栏中的最后一个名称为Volume Visualization的按钮。此时会弹出一个界面,用来设置体渲染的一些选项。
勾选第一项Volume Rendering。则在第四个窗口中会显示数据体三维重建,并且增加了预设的颜色和透明度传递函数后的结果。

3. 基于MITK的区域生长分割:
由于MITK集成了ITK和VTK。在ITK中提供了很多成熟的图像分割和配准的算法。因此,MITK将ITK封装的这些算法提供了出来。以下简要介绍其中的区域生长分割算法的应用。点击工具栏从右往左数第四个,名称为Segmentation的按钮,即为图像分割的按钮。
此时,会弹出一个图像分割的工具箱面板。
面板的最上面部分为Data Selection,是用户选择待分割的图像和创建保存分割结果的。
默认将体数据作为分割对象。创建一个名称为bone_segment的标签,作为保存分割结果的文件。
该分割工具箱面板提供了分割的2D Tool和3D Tool。
2D Tool包括:Add, Subtract, Correction, Paint, Wipe, Region Growing, Fill, Erase, Live Wire, 2D Fast Marching的分割功能;
3D Tool包括:Threshold, UL Threshold, Otsu, Fast Marching 3D, Region Growing 3D, Watershed, Picking的分割功能。
这里介绍Region Growing 3D算法。

Region Growing在ITK中有不同的实现算法。通过这里的界面显示,MITK中应用的应该是ITK中的connectedThresholdImageFilter的类来实现区域生长的分割功能。
默认的阈值上下限为体数据的灰度最小值和最大值。这里载入的是CT数据,所以下限是-1024.0, 上限是1791.0。通过查阅资料,要分割的股骨区域的灰度范围约为280~1791,因此修改下限值为280。
然后点击键盘上的Shift键后,同时用鼠标在MPR的任一个面上选取股骨区域的一个点作为种子点。如下图所示为,在冠状位上选取了一个种子点。

然后点击分割面板中的“Run Segmentation”按钮,即可以在MPR区域显示出区域生长后的区域。选中分割面板的3D preview选项,则会将分割后的区域进行三维重建,显示在第四个窗口中。

参考链接:
1. http://mitk.org/wiki/MITK
2. http://www.mitk.net/
3. http://www.oschina.net/p/mitk
相关文章推荐
- 通过web升级HF wifi固件
- 数据库设计的一些心得与经验
- 什么是原生的javascript
- Codeforces 494D Upgrading Array
- SharpZipLib.dll 压缩文件,可以应用于MVC, webform. C# windows application 等等地方
- 双向循环链表
- 手把手教你使用Git--Git入门
- 数据库设计准则(第一、第二、第三范式说明)
- JavaScript跨域总结与解决办法
- 类的加载到反射reflect
- Maximal Square
- [JZOJ3430] DY引擎
- Activity创建流程
- ThinkPHP框架表单验证
- Cordova VS React Native 谁是未来? - b
- 第二章:probability distribution
- 71. Simplify Path
- Centos Linux系统下配置网站开发环境(jdk7+mysql5.6+tomcat)
- java做微信支付notify_url异步通知服务端的写法
- Android JSBridge实现与前端的交互
